Das helfRlein Package – Eine Sammlung nützlicher Funktionen


Um diese Funktionen innerhalb unserer Teams und auch mit anderen zu teilen, habe ich angefangen, sie zu sammeln und habe dann daraus ein R-Paket namens helfRlein erstellt. Neben der gemeinsamen Nutzung wollte ich auch einige Anwendungsfälle haben, um meine Fähigkeiten zur Fehlersuche und Optimierung zu verbessern. Mit der Zeit wuchs das Paket und es kamen immer mehr Funktionen zusammen. Beim letzten Mal habe ich jede Funktion als Teil eines Adventskalenders vorgestellt. Zum Start unserer neuen Website habe ich alle Funktionen in diesem Kalender zusammengefasst und werde jede aktuelle Funktion aus dem Paket helfRlein vorstellen.
Die meisten Funktionen wurden entwickelt, als es ein Problem gab und man eine einfache Lösung dafür brauchte. Zum Beispiel war der angezeigte Text zu lang und musste gekürzt werden (siehe evenstrings). Andere Funktionen existieren nur, um sich-wiederholende Aufgaben zu reduzieren – wie das Einlesen mehrerer Dateien des selben Typs (siehe read_files). Daher könnten diese Funktionen auch für Euch nützlich sein!
Um alle Funktionen im Detail zu erkunden, könnt Ihr unser GitHub besuchen. Wenn Ihr irgendwelche Vorschläge habt, schickt mir bitte eine E-Mail oder öffnet ein Issue auf GitHub!
1. char_replace
Dieser kleine Helfer ersetzt Sonderzeichen (wie z. B. den Umlaut "ä") durch ihre Standardentsprechung (in diesem Fall "ae"). Es ist auch möglich, alle Zeichen in Kleinbuchstaben umzuwandeln, Leerzeichen zu entfernen oder Leerzeichen und Bindestriche durch Unterstriche zu ersetzen.
Schauen wir uns ein kleines Beispiel mit verschiedenen Settings an:
x <- " Élizàldë-González Strasse"
char_replace(x, to_lower = TRUE)[1] "elizalde-gonzalez strasse"char_replace(x, to_lower = TRUE, to_underscore = TRUE)[1] "elizalde_gonzalez_strasse"char_replace(x, to_lower = FALSE, rm_space = TRUE, rm_dash = TRUE)[1] "ElizaldeGonzalezStrasse"
2. checkdir
Dieser kleine Helfer prüft einen gegebenen Ordnerpfad auf Existenz und erstellt ihn bei Bedarf.
checkdir(path = "testfolder/subfolder")
Intern gibt es nur eine einfache if-Anweisung, die die R-Basisfunktionen file.exists() und dir.create(). kombiniert.
3. clean_gc
Dieser kleine Helfer gibt den Speicher von unbenutzten Objekten frei. Nun, im Grunde ruft es einfach gc() ein paar Mal auf. Ich habe das vor einiger Zeit für ein Projekt benutzt, bei dem ich mit riesigen Datendateien gearbeitet habe. Obwohl wir das Glück hatten, einen großen Server mit 500 GB RAM zu haben, stießen wir bald an seine Grenzen. Da wir in der Regel mehrere Prozesse parallelisieren, mussten wir jedes Bit und jedes Byte des Arbeitsspeichers nutzen, das wir bekommen konnten. Anstatt also viele Zeilen wie diese zu haben:
gc();gc();gc();gc()
... habe ich clean_gc() der Einfachheit halber geschrieben. Intern wird gc() so lange aufgerufen, wie es Speicher gibt, der freigegeben werden muss.
Some further thoughts
Es gibt einige Diskussionen über den Garbage Collector gc() und seine Nützlichkeit. Wenn Ihr mehr darüber erfahren wollt, schlage ich vor, dass Ihr Euch die memory section in Advanced R anseht. Ich weiß, dass R selbst bei Bedarf Speicher freigibt, aber ich bin mir nicht sicher, was passiert, wenn Ihr mehrere R-Prozesse habt. Können sie den Speicher von anderen Prozessen leeren? Wenn Ihr dazu etwas mehr wisst, lasst es mich wissen!
4. count_na
Dieser kleine Helfer zählt fehlende Werte innerhalb eines Vektors.
x <- c(NA, NA, 1, NaN, 0)
count_na(x)3
Intern gibt es nur ein einfaches sum(is.na(x)), das die NA-Werte zählt. Wenn Ihr den Mittelwert statt der Summe wollt, könnt Ihr prop = TRUE setzen.
5. evenstrings
Dieser kleine Helfer zerlegt eine gegebene Zeichenkette in kleinere Teile mit einer festen Länge. Aber warum? Nun, ich brauchte diese Funktion beim Erstellen eines Plots mit einem langen Titel. Der Text war zu lang für eine Zeile und anstatt ihn einfach abzuschneiden oder über die Ränder laufen zu lassen, wollte ich ihn schön trennen.
Bei einer langen Zeichenkette wie...
long_title <- c("Contains the months: January, February, March, April, May, June, July, August, September, October, November, December")
...wollen wir sie nach split = "," mit einer maximalen Länge von char = 60 aufteilen.
short_title <- evenstrings(long_title, split = ",", char = 60)
Die Funktion hat zwei mögliche Ausgabeformate, die durch Setzen von newlines = TRUE oder FALSE gewählt werden können:
- eine Zeichenkette mit Zeilentrennzeichen
\n - ein Vektor mit jedem Unterteil.
Ein anderer Anwendungsfall könnte eine Nachricht sein, die mit cat() auf der Konsole ausgegeben wird:
cat(long_title)Contains the months: January, February, March, April, May, June, July, August, September, October, November, Decembercat(short_title)Contains the months: January, February, March, April, May,
June, July, August, September, October, November, December
Code for plot example
p1 <- ggplot(data.frame(x = 1:10, y = 1:10),
aes(x = x, y = y)) +
geom_point() +
ggtitle(long_title)
p2 <- ggplot(data.frame(x = 1:10, y = 1:10),
aes(x = x, y = y)) +
geom_point() +
ggtitle(short_title)
multiplot(p1, p2)
6. get_files
Dieser kleine Helfer macht das Gleiche wie die "Find in files " Suche in RStudio. Sie gibt einen Vektor mit allen Dateien in einem bestimmten Ordner zurück, die das Suchmuster enthalten. In Eurem täglichen Arbeitsablauf würdet Ihr normalerweise die Tastenkombination SHIFT+CTRL+F verwenden. Mit get_files() könnt Ihr diese Funktionen in Euren Skripten nutzen.
7. get_network
Das Ziel dieses kleinen Helfers ist es, die Verbindungen zwischen R-Funktionen innerhalb eines Projekts als Flussdiagramm zu visualisieren. Dazu ist die Eingabe ein Verzeichnispfad zur Funktion oder eine Liste mit den Funktionen und die Ausgaben sind eine Adjazenzmatrix und ein Graph-Objekt. Als Beispiel verwenden wir diesen Ordner mit einigen Spielzeugfunktionen:
net <- get_network(dir = "flowchart/R_network_functions/", simplify = FALSE)
g1 <- net(dollar sign)igraph
Input
Es gibt fünf Parameter, um mit der Funktion zu interagieren:
- ein Pfad
dir, der durchsucht werden soll. - ein Zeichenvektor
Variationenmit der Definitionszeichenfolge der Funktion – die Vorgabe istc(" <- function", "<- function", "<-function"). - ein “Muster”, eine Zeichenkette mit dem Dateisuffix – die Vorgabe ist
"\\.R (dollar sign)" - ein boolesches
simplify, das Funktionen ohne Verbindungen aus der Darstellung entfernt. - eine benannte Liste
all_scripts, die eine Alternative zudirist. Diese Liste wird hauptsächlich nur zu Testzwecken verwendet.
Für eine normale Verwendung sollte es ausreichen, einen Pfad zum Projektordner anzugeben.
Output
Der gegebene Plot zeigt die Verbindungen der einzelnen Funktionen (Pfeile) und auch die relative Größe des Funktionscodes (Größe der Punkte). Wie bereits erwähnt, besteht die Ausgabe aus einer Adjazenzmatrix und einem Graph-Objekt. Die Matrix enthält die Anzahl der Aufrufe für jede Funktion. Das Graph-Objekt hat die folgenden Eigenschaften:
- Die Namen der Funktionen werden als Label verwendet.
- Die Anzahl der Zeilen jeder Funktion (ohne Kommentare und Leerzeilen) wird als Größe gespeichert.
- Der Ordnername des ersten Ordners im Verzeichnis.
- Eine Farbe, die dem Ordner entspricht.
Mit diesen Eigenschaften können Sie die Netzwerkdarstellung zum Beispiel wie folgt verbessern:
library(igraph)
# create plots ------------------------------------------------------------
l <- layout_with_fr(g1)
colrs <- rainbow(length(unique(V(g1)(dollar sign)color)))
plot(g1,
edge.arrow.size = .1,
edge.width = 5*E(g1)(dollar sign)weight/max(E(g1)(dollar sign)weight),
vertex.shape = "none",
vertex.label.color = colrs[V(g1)(dollar sign)color],
vertex.label.color = "black",
vertex.size = 20,
vertex.color = colrs[V(g1)(dollar sign)color],
edge.color = "steelblue1",
layout = l)
legend(x = 0,
unique(V(g1)(dollar sign)folder), pch = 21,
pt.bg = colrs[unique(V(g1)(dollar sign)color)],
pt.cex = 2, cex = .8, bty = "n", ncol = 1)
8. get_sequence
Dieser kleine Helfer gibt Indizes von wiederkehrenden Mustern zurück. Es funktioniert sowohl mit Zahlen als auch mit Zeichen. Alles, was es braucht, ist ein Vektor mit den Daten, ein Muster, nach dem gesucht werden soll, und eine Mindestanzahl von Vorkommen.
Lasst uns mit dem folgenden Code einige Zeitreihendaten erstellen.
library(data.table)
# random seed
set.seed(20181221)
# number of observations
n <- 100
# simulationg the data
ts_data <- data.table(DAY = 1:n, CHANGE = sample(c(-1, 0, 1), n, replace = TRUE))
ts_data[, VALUE := cumsum(CHANGE)]
Dies ist nichts anderes als ein Random Walk, da wir zwischen dem Abstieg (-1), dem Anstieg (1) und dem Verbleib auf demselben Niveau (0) wählen. Unsere Zeitreihendaten sehen folgendermaßen aus:
Angenommen, wir wollen die Datumsbereiche wissen, in denen es an mindestens vier aufeinanderfolgenden Tagen keine Veränderung gab.
ts_data[, get_sequence(x = CHANGE, pattern = 0, minsize = 4)] min max
[1,] 45 48
[2,] 65 69
Wir können auch die Frage beantworten, ob sich das Muster "down-up-down-up" irgendwo wiederholt:
ts_data[, get_sequence(x = CHANGE, pattern = c(-1,1), minsize = 2)] min max
[1,] 88 91
Mit diesen beiden Eingaben können wir unseren Plot ein wenig aktualisieren, indem wir etwas geom_rect hinzufügen!

Code for the plot
rect <- data.table(
rbind(ts_data[, get_sequence(x = CHANGE, pattern = c(0), minsize = 4)],
ts_data[, get_sequence(x = CHANGE, pattern = c(-1,1), minsize = 2)]),
GROUP = c("no change","no change","down-up"))
ggplot(ts_data, aes(x = DAY, y = VALUE)) +
geom_line() +
geom_rect(data = rect,
inherit.aes = FALSE,
aes(xmin = min - 1,
xmax = max,
ymin = -Inf,
ymax = Inf,
group = GROUP,
fill = GROUP),
color = "transparent",
alpha = 0.5) +
scale_fill_manual(values = statworx_palette(number = 2, basecolors = c(2,5))) +
theme_minimal()9. intersect2
Dieser kleine Helfer gibt den Schnittpunkt mehrerer Vektoren oder Listen zurück. Ich habe diese Funktion hier gefunden, fand sie recht nützlich und habe sie ein wenig angepasst.
intersect2(list(c(1:3), c(1:4)), list(c(1:2),c(1:3)), c(1:2))[1] 1 2
Intern wird das Problem, die Schnittmenge zu finden, rekursiv gelöst, wenn ein Element eine Liste ist, und dann schrittweise mit dem nächsten Element.
10. multiplot
Dieses kleine Hilfsmittel kombiniert mehrere ggplots zu einem Plot. Dies ist eine Funktion aus dem R-cookbook.
Ein Vorteil gegenüber facets ist, dass man nicht alle Daten für alle Plots in einem Objekt benötigt. Auch kann man jeden einzelnen Plot frei erstellen – was manchmal auch ein Nachteil sein kann.
Mit dem Parameter layout könnt Ihr mehrere Plots mit unterschiedlichen Größen anordnen. Nehmen wir an, Ihr habt drei Plots und wollt sie wie folgt anordnen:
1 2 2
1 2 2
3 3 3Bei multiplot läuft es auf Folgendes hinaus:
multiplot(plotlist = list(p1, p2, p3),
layout = matrix(c(1,2,2,1,2,2,3,3,3), nrow = 3, byrow = TRUE))
Code for plot example
# star coordinates
c1 = cos((2*pi)/5)
c2 = cos(pi/5)
s1 = sin((2*pi)/5)
s2 = sin((4*pi)/5)
data_star <- data.table(X = c(0, -s2, s1, -s1, s2),
Y = c(1, -c2, c1, c1, -c2))
p1 <- ggplot(data_star, aes(x = X, y = Y)) +
geom_polygon(fill = "gold") +
theme_void()
# tree
set.seed(24122018)
n <- 10000
lambda <- 2
data_tree <- data.table(X = c(rpois(n, lambda), rpois(n, 1.1*lambda)),
TYPE = rep(c("1", "2"), each = n))
data_tree <- data_tree[, list(COUNT = .N), by = c("TYPE", "X")]
data_tree[TYPE == "1", COUNT := -COUNT]
p2 <- ggplot(data_tree, aes(x = X, y = COUNT, fill = TYPE)) +
geom_bar(stat = "identity") +
scale_fill_manual(values = c("green", "darkgreen")) +
coord_flip() +
theme_minimal()
# gifts
data_gifts <- data.table(X = runif(5, min = 0, max = 10),
Y = runif(5, max = 0.5),
Z = sample(letters[1:5], 5, replace = FALSE))
p3 <- ggplot(data_gifts, aes(x = X, y = Y)) +
geom_point(aes(color = Z), pch = 15, size = 10) +
scale_color_brewer(palette = "Reds") +
geom_point(pch = 12, size = 10, color = "gold") +
xlim(0,8) +
ylim(0.1,0.5) +
theme_minimal() +
theme(legend.position="none")
11. na_omitlist
Dieser kleine Helfer entfernt fehlende Werte aus einer Liste.
y <- list(NA, c(1, NA), list(c(5:6, NA), NA, "A"))
Es gibt zwei Möglichkeiten, die fehlenden Werte zu entfernen, entweder nur auf der ersten Ebene der Liste oder innerhalb jeder Unterebene.
na_omitlist(y, recursive = FALSE)[[1]]
[1] 1 NA
[[2]]
[[2]][[1]]
[1] 5 6 NA
[[2]][[2]]
[1] NA
[[2]][[3]]
[1] "A"na_omitlist(y, recursive = TRUE)[[1]]
[1] 1
[[2]]
[[2]][[1]]
[1] 5 6
[[2]][[2]]
[1] "A"
12. %nin%
Dieser kleine Helfer ist eine reine Komfortfunktion. Sie ist einfach dasselbe wie der negierte %in%-Operator, wie Ihr unten sehen könnt. Aber meiner Meinung nach erhöht sie die Lesbarkeit des Codes.
all.equal( c(1,2,3,4) %nin% c(1,2,5),
!c(1,2,3,4) %in% c(1,2,5))[1] TRUE
Dieser Operator hat es auch in einige andere Pakete geschafft – wie Ihr hier nachlesen könnt.
13. object_size_in_env
Dieser kleine Helfer zeigt eine Tabelle mit der Größe jedes Objekts in der vorgegebenen Umgebung an.
Wenn Ihr in einer Situation seid, in der Ihr viel gecodet habt und Eure Umgebung nun ziemlich unübersichtlich ist, hilft Euch object_size_in_env, die großen Fische in Bezug auf den Speicherverbrauch zu finden. Ich selbst bin ein paar Mal auf dieses Problem gestoßen, als ich mehrere Ausführungen meiner Modelle in einem Loop durchlaufen habe. Irgendwann wurden die Sitzungen ziemlich groß im Speicher und ich wusste nicht, warum! Mit Hilfe von object_size_in_env und etwas Degubbing konnte ich das Objekt ausfindig machen, das dieses Problem verursachte, und meinen Code entsprechend anpassen.
Zuerst wollen wir eine Umgebung mit einigen Variablen erstellen.
# building an environment
this_env <- new.env()
assign("Var1", 3, envir = this_env)
assign("Var2", 1:1000, envir = this_env)
assign("Var3", rep("test", 1000), envir = this_env)
Um die Größeninformationen unserer Objekte zu erhalten, wird intern format(object.size()) verwendet. Mit der Einheit kann das Ausgabeformat geändert werden (z.B. "B", "MB" oder "GB").
# checking the size
object_size_in_env(env = this_env, unit = "B") OBJECT SIZE UNIT
1: Var3 8104 B
2: Var2 4048 B
3: Var1 56 B
14. print_fs
Dieser kleine Helfer gibt die Ordnerstruktur eines gegebenen Pfades zurück. Damit kann man z.B. eine schöne Übersicht in die Dokumentation eines Projektes oder in ein Git einbauen. Im Sinne der Automatisierung könnte diese Funktion nach einer größeren Änderung Teile in einer Log- oder News-Datei ändern.
Wenn wir uns das gleiche Beispiel anschauen, das wir für die Funktion get_network verwendet haben, erhalten wir folgendes:
print_fs("~/flowchart/", depth = 4)1 flowchart
2 ¦--create_network.R
3 ¦--getnetwork.R
4 ¦--plots
5 ¦ ¦--example-network-helfRlein.png
6 ¦ °--improved-network.png
7 ¦--R_network_functions
8 ¦ ¦--dataprep
9 ¦ ¦ °--foo_01.R
10 ¦ ¦--method
11 ¦ ¦ °--foo_02.R
12 ¦ ¦--script_01.R
13 ¦ °--script_02.R
14 °--README.md
Mit depth können wir einstellen, wie tief wir unsere Ordner durchforsten wollen.
15. read_files
Dieser kleine Helfer liest mehrere Dateien des selben Typs ein und fasst sie zu einer data.table zusammen. Welche Art von Dateilesefunktion verwendet werden soll, kann mit dem Argument FUN ausgewählt werden.
Wenn Sie eine Liste von Dateien haben, die alle mit der gleichen Funktion eingelesen werden sollen (z.B. read.csv), können Sie statt lapply und rbindlist nun dies verwenden:
read_files(files, FUN = readRDS)
read_files(files, FUN = readLines)
read_files(files, FUN = read.csv, sep = ";")
Intern verwendet es nur lapply und rbindlist, aber man muss es nicht ständig eingeben. Die read_files kombiniert die einzelnen Dateien nach ihren Spaltennamen und gibt eine data.table zurück. Warum data.table? Weil ich es mag. Aber lassen Sie uns nicht das Fass von data.table vs. dplyr aufmachen (zum Fass…).
16. save_rds_archive
Dieser kleine Helfer ist ein Wrapper um die Basis-R-Funktion saveRDS() und prüft, ob die Datei, die Ihr zu speichern versucht, bereits existiert. Wenn ja, wird die bestehende Datei umbenannt / archiviert (mit einem Zeitstempel), und die "aktualisierte" Datei wird unter dem angegebenen Namen gespeichert. Das bedeutet, dass vorhandener Code, der davon abhängt, dass der Dateiname konstant bleibt (z.B. readRDS()-Aufrufe in anderen Skripten), weiterhin funktionieren wird, während eine archivierte Kopie der – ansonsten überschriebenen – Datei erhalten bleibt.
17. sci_palette
Dieser kleine Helfer liefert eine Reihe von Farben, die wir bei statworx häufig verwenden. Wenn Ihr Euch also - so wie ich - nicht an jeden Hex-Farbcode erinnern könnt, den Ihr braucht, könnte das helfen. Natürlich sind das unsere Farben, aber Ihr könnt es auch mit Eurer eigenen Farbpalette umschreiben. Aber der Hauptvorteil ist die Plot-Methode – so könnt Ihr die Farbe sehen, anstatt nur den Hex-Code zu lesen.
So seht Ihr, welcher Hexadezimalcode welcher Farbe entspricht und wofür Ihr ihn verwenden könnt.
sci_palette(scheme = "new")Tech Blue Black White Light Grey Accent 1 Accent 2 Accent 3
"#0000FF" "#000000" "#FFFFFF" "#EBF0F2" "#283440" "#6C7D8C" "#B6BDCC"
Highlight 1 Highlight 2 Highlight 3
"#00C800" "#FFFF00" "#FE0D6C"
attr(,"class")
[1] "sci"Wie bereits erwähnt, gibt es eine Methode plot(), die das folgende Bild ergibt.
plot(sci_palette(scheme = "new"))
18. statusbar
Dieser kleine Helfer gibt einen Fortschrittsbalken in der Konsole für Schleifen aus.
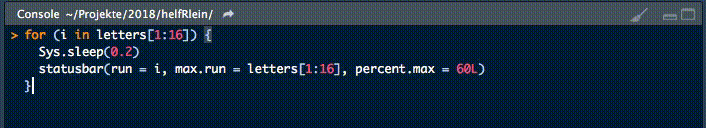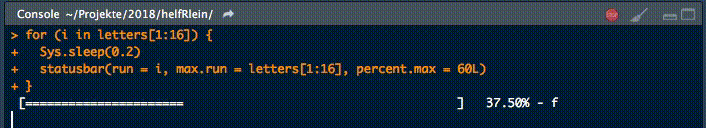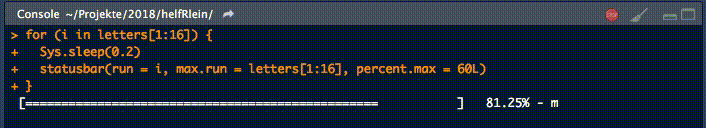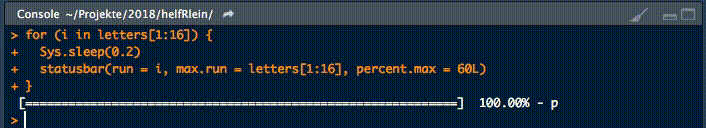
Es gibt zwei notwendige Parameter, um diese Funktion zu füttern:
runist entweder der Iterator oder seine Nummermax.runist entweder alle möglichen Iteratoren in der Reihenfolge, in der sie verarbeitet werden, oder die maximale Anzahl von Iterationen.
So könnte es zum Beispiel run = 3 und max.run = 16 oder run = "a" und max.run = Buchstaben[1:16] sein.
Außerdem gibt es zwei optionale Parameter:
percent.maxbeeinflusst die Breite des Fortschrittsbalkensinfoist ein zusätzliches Zeichen, das am Ende der Zeile ausgegeben wird. Standardmäßig ist esrun.
Ein kleiner Nachteil dieser Funktion ist, dass sie nicht mit parallelen Prozessen arbeitet. Wenn Ihr einen Fortschrittsbalken haben wollt, wenn Ihr apply Funktionen benutzt, schaut Euch pbapply an.
19. statworx_palette
Dieses kleine Hilfsmittel ist eine Ergänzung zu sci_palette(). Wir haben die Farben 1, 2, 3, 5 und 10 ausgewählt, um eine flexible Farbpalette zu erstellen. Wenn Sie 100 verschiedene Farben benötigen - sagen Sie nichts mehr!
Im Gegensatz zu sci_palette() ist der Rückgabewert ein Zeichenvektor. Zum Beispiel, wenn Sie 16 Farben wollen:
statworx_palette(16, scheme = "old")[1] "#013848" "#004C63" "#00617E" "#00759A" "#0087AB" "#008F9C" "#00978E" "#009F7F"
[9] "#219E68" "#659448" "#A98B28" "#ED8208" "#F36F0F" "#E45A23" "#D54437" "#C62F4B"
Wenn wir nun diese Farben aufzeichnen, erhalten wir einen schönen regenbogenartigen Farbverlauf.
library(ggplot2)
ggplot(plot_data, aes(x = X, y = Y)) +
geom_point(pch = 16, size = 15, color = statworx_palette(16, scheme = "old")) +
theme_minimal()
Eine zusätzliche Funktion ist der Parameter reorder, der die Reihenfolge der Farben abtastet, so dass Nachbarn vielleicht etwas besser unterscheidbar sind. Auch wenn Sie die verwendeten Farben ändern wollen, können Sie dies mit basecolors tun.
ggplot(plot_data, aes(x = X, y = Y)) +
geom_point(pch = 16, size = 15,
color = statworx_palette(16, basecolors = c(4,8,10), scheme = "new")) +
theme_minimal()
20. strsplit
Dieses kleine Hilfsmittel erweitert die R-Basisfunktion strsplit – daher der gleiche Name! Es ist nun möglich, before, after oder between ein bestimmtes Begrenzungszeichen zu trennen. Im Falle von between müsst ihr zwei Delimiter angeben.
Hier ist ein kleines Beispiel, wie man das neue strsplit benutzt.
text <- c("This sentence should be split between should and be.")
strsplit(x = text, split = " ")
strsplit(x = text, split = c("should", " be"), type = "between")
strsplit(x = text, split = "be", type = "before")[[1]]
[1] "This" "sentence" "should" "be" "split" "between" "should" "and"
[9] "be."
[[1]]
[1] "This sentence should" " be split between should and be."
[[1]]
[1] "This sentence should " "be split " "between should and "
[4] "be."
21. to_na
Dieser kleine Helfer ist nur eine Komfortfunktion. Bei der Datenaufbereitung kann es vorkommen, dass Ihr einen Vektor mit unendlichen Werten wie Inf oder -Inf oder sogar NaN-Werten habt. Solche Werte können (müssen aber nicht!) Eure Auswertungen und Modelle durcheinanderbringen. Aber die meisten Funktionen haben die Tendenz, fehlende Werte zu behandeln. Daher entfernt diese kleine Hilfe solche Werte und ersetzt sie durch NA.
Ein kleines Beispiel, um Euch die Idee zu vermitteln:
test <- list(a = c("a", "b", NA),
b = c(NaN, 1,2, -Inf),
c = c(TRUE, FALSE, NaN, Inf))
lapply(test, to_na)(dollar sign)a
[1] "a" "b" NA
(dollar sign)b
[1] NA 1 2 NA
(dollar sign)c
[1] TRUE FALSE NA
Ein kleiner Tipp am Rande! Da es je nach den anderen Werten innerhalb eines Vektors verschiedene Arten von NA gibt, solltet Ihr das Format überprüfen, wenn Ihr to_na auf Gruppen oder Teilmengen anwendet.
test <- list(NA, c(NA, "a"), c(NA, 2.3), c(NA, 1L))
str(test)List of 4
(dollar sign) : logi NA
(dollar sign) : chr [1:2] NA "a"
(dollar sign) : num [1:2] NA 2.3
(dollar sign) : int [1:2] NA 1
22. trim
Dieser kleine Helfer entfernt führende und nachfolgende Leerzeichen aus einer Zeichenkette. Mit R Version 3.5.1 wurde trimws eingeführt, das genau das Gleiche tut. Das zeigt nur, dass es keine schlechte Idee war, eine solche Funktion zu schreiben. 😉
x <- c(" Hello world!", " Hello world! ", "Hello world! ")
trim(x, lead = TRUE, trail = TRUE)[1] "Hello world!" "Hello world!" "Hello world!
Die Parameter lead und trail geben an, ob nur die führenden, die nachfolgenden oder beide Leerzeichen entfernt werden sollen.
Fazit
Ich hoffe, dass euch das helfRlein Package genauso die Arbeit erleichtert, wie uns hier bei statworx. Schreibt uns bei Fragen oder Input zum Package gerne eine Mail an: blog@statworx.com


