Wie Du ein R-Skript in Docker ausführst


Seit der Veröffentlichung im Jahr 2014 hat sich Docker zu einem unverzichtbaren Werkzeug für die Bereitstellung von Anwendungen entwickelt. Bei statworx ist R eines unserer täglichen Werkzeuge, weshalb viele von uns begeistert sind von RStudio's Rocker Projekt, das die Containerisierung von R-Code einfacher macht denn je.Containerisierung ist in vielen verschiedenen Situationen nützlich. Die Technologie ist äußerst hilfreich beim Einsatz von R-Code in einer Cloud-Computing-Umgebung, in welcher der gecodete Arbeitsablauf in regelmäßigen Abständen ausgeführt werden soll. Docker ist für diese Aufgabe aus zwei Gründen perfekt geeignet: Container können automatisiert in gewünschten Intervallen gestartet werden und aufgrund ihrer statischen Natur ist immer klar, welches Verhalten und welchen Output du von einem Docker-Container zu erwarten hast. Wenn du also vor der Aufgabe stehst, ein Machine-Learning-Modell für regelmäßige Prognosen einzusetzen, dann lohnt es sich dies mit Docker tun.
Dieser Blogbeitrag führt dich Schritt für Schritt durch den gesamten Prozess, wie du dein R-Skript in einem Docker-Container zum Laufen bringst. Der Einfachheit halber werden wir mit einem lokalen Datensatz arbeiten.Zu Beginn möchte ich betonen, dass dieser Blogbeitrag kein allgemeines Docker-Tutorial ist. Wenn du dir nicht sicher bist was mit Images und Containern gemeint ist, dann empfehle ich dir, zunächst einen Blick auf das Docker Curriculum zu werfen. Solltest du daran interessiert sein, eine RStudio-Sitzung in einem Docker-Container laufen zu lassen, empfehle ich dir stattdessen dem OpenSciLabs Docker Tutorial einen Besuch abzustatten.In diesem Blogbeitrag geht es konkret um die Containerisierung eines R-Skripts, damit es schließlich bei jedem Start des Containers automatisch ausgeführt wird, ohne dass der Benutzer eingreifen muss – damit entfällt die Notwendigkeit des RStudio-IDE. Ich werde nur kurz auf die im Dockerfile und in der Kommandozeile verwendete Syntax eingehen, so dass du dich am besten mit den Grundlagen von Docker bereits vor dem Weiterlesen vertraut machst.
Was wir brauchen:
Für diesen gesamten Workflow benötigen wir folgendes:
- ein R-Skript, das wir in ein Docker-Image einbauen
- ein Base-Image, auf dem wir unser eigenes Image aufbauen
- ein Dockerfile, mit der wir unser neues Image definieren
Du kannst alle Dateien und die verwendete Ordnerstruktur aus dem statworx GitHub Repository klonen.
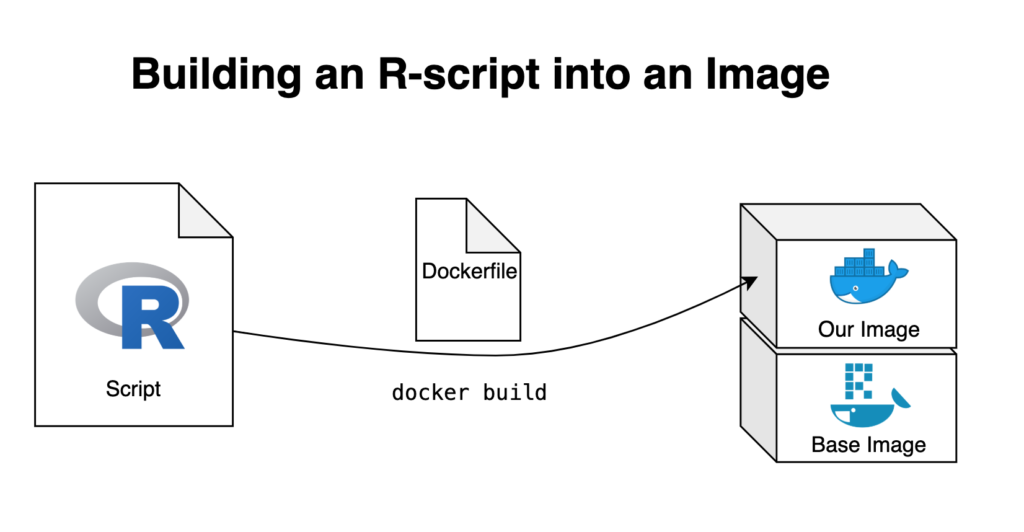
Das R-Skript
Wir arbeiten mit einem sehr einfachen R-Skript, das einen Datensatz importiert, als Dataframe manipuliert, ein Diagramm auf der Grundlage der manipulierten Daten erstellt und zum Schluss sowohl das Diagramm als auch die darauf basierenden Daten exportiert. Der für dieses Beispiel verwendete Datensatz ist US 500 Records, der von Brian Dunning zur Verfügung gestellt wird. Wenn du dem Beispiel folgen möchtest, empfehle ich dir, diesen Datensatz in den Ordner 01_data zu kopieren.
library(readr)
library(dplyr)
library(ggplot2)
library(forcats)
# import dataframe
df <- read_csv("01_data/us-500.csv")
# manipulate data
plot_data <- df %>%
group_by(state) %>%
count()
# save manipulated data to output folder
write_csv(plot_data, "03_output/plot_data.csv")
# create plot based on manipulated data
plot <- plot_data %>%
ggplot()+
geom_col(aes(fct_reorder(state, n),
n,
fill = n))+
coord_flip()+
labs(
title = "Number of people by state",
subtitle = "From US-500 dataset",
x = "State",
y = "Number of people"
)+
theme_bw()
# save plot to output folder
ggsave("03_output/myplot.png", width = 10, height = 8, dpi = 100)
Damit wird ein einfaches Balkendiagramm auf der Grundlage des Datensatzes erstellt:
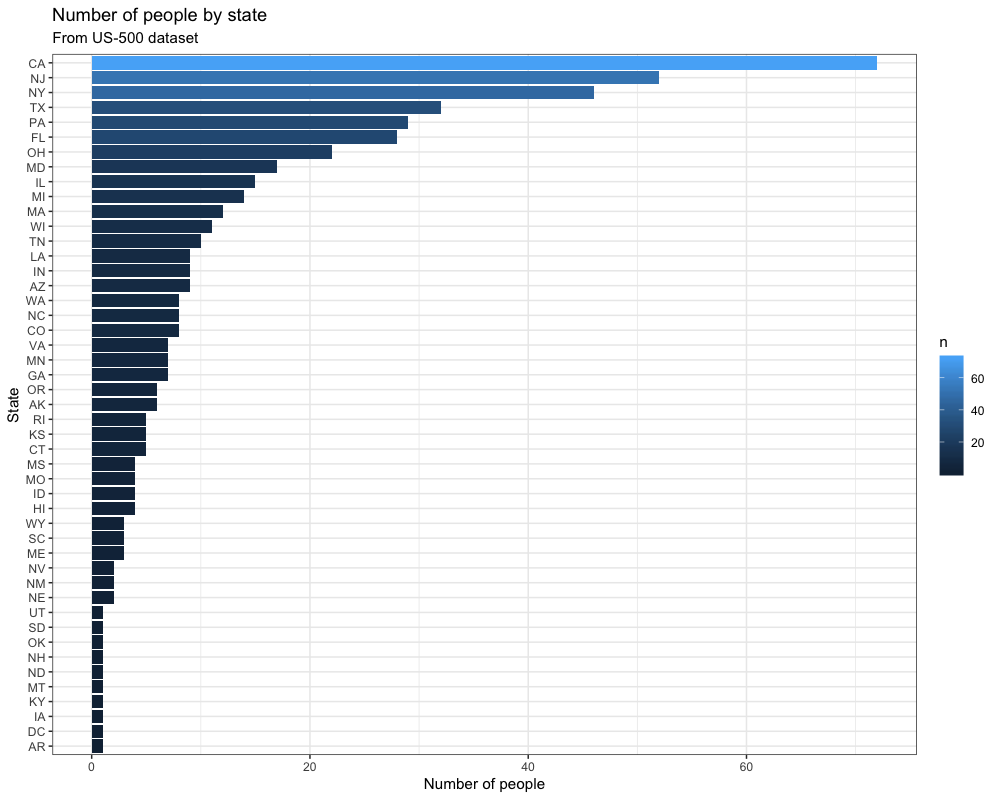
Wir verwenden dieses Skript, weil wir nicht nur R-Code innerhalb eines Docker-Containers ausführen, sondern diesen R-Code auf Daten von außerhalb unseres Containers anwenden und die Ergebnisse anschließend speichern wollen.
Das Base-Image
Die DockerHub-Seite des Rocker-Projekts listet alle verfügbaren Rocker-Repositories auf. Da wir in unserem Skript Tidyverse-Pakete verwenden, ist das rocker/tidyverse-Image eine naheliegende Wahl. Das Problem mit diesem Repository ist, dass es auch RStudio selbst enthält, was wir für dieses spezifische Projekt nicht benötigen. Das bedeutet, dass wir stattdessen mit dem r-base Repository arbeiten und unser eigenes Tidyverse-fähiges Image erstellen. Wir können das rocker/r-base-Image von DockerHub nutzen, indem wir den folgenden Befehl im Terminal ausführen:
docker pull rocker/r-base
Dadurch wird das Base-R-Image aus dem Rocker DockerHub-Repository lokal gespeichert ("gepullt"). Wir können einen auf diesem Image basierenden Container starten, indem wir im Terminal folgenden Bashcode ausführen:
docker run -it --rm rocker/r-base
Herzlichen Glückwunsch, du führst jetzt R innerhalb eines Docker-Containers aus! Das Terminal wurde in eine R-Konsole verwandelt, mit der wir dank des Arguments -it interagieren können. Das Argument --rm sorgt dafür, dass der Container automatisch gelöscht wird, sobald wir ihn stoppen. Es steht dir frei, mit deiner containerisierten R-Sitzung zu experimentieren (die du mit der Funktion q() in der R-Konsole wieder beenden kannst). Du kannst zum Beispiel damit beginnen, die Pakete, die du für deinen Arbeitsablauf benötigst, mit install.packages() zu installieren, aber das ist eine mühsame und zeitraubende Herangehensweise. Besser ist es, die gewünschten Pakete bereits in das Image einzubauen, damit die benötigten Pakete nicht nach jedem Containerstart erneut manuell installiert werden müssen. Dazu benötigen wir ein Dockerfile.
Das Dockerfile
Mit einem Dockerfile teilen wir Docker mit, wie unser gewünschtes Image erstellt werden soll. Ein Dockerfile ist eine Textdatei, die "Dockerfile.txt" heißen muss und standardmäßig im Stammverzeichnis des "Build-Kontextes" liegt (in unserem Fall wäre das der Ordner "R-Script in Docker").
Zunächst legen wir ein bestehendes Docker-Image fest, basierend auf dem wir unser neues Image erstellen möchten. Anschließend verfassen wir eine Liste von Anweisungen, die unser Image so definieren, dass die Ausführung von Containern reibungslos und effizient verläuft. In unserem Fall möchte ich unser neues Image auf dem zuvor besprochenen rocker/r-base-Image aufbauen. Ebenfalls möchte ich auch die lokale Ordnerstruktur replizieren, also erstelle ich die gewünschten Verzeichnisse direkt mit dem Dockerfile. Danach werden alle Dateien, auf die das Image Zugriff haben soll, in diese Verzeichnisse kopiert – so wird das R-Skript in das Docker-Image eingebaut. Auf diese Weise können wir auch die manuelle Installation von Paketen nach dem Starten eines Containers umgehen, da wir ein zweites R-Skript vorbereiten können, das sich um die Paketinstallation kümmert. Es reicht nicht aus, das R-Skript einfach zu kopieren, wir müssen Docker auch anweisen, es beim Erstellen des Images automatisch auszuführen. Und das ist unser erstes Dockerfile:
# Base image https://hub.docker.com/u/rocker/
FROM rocker/r-base:latest
## create directories
RUN mkdir -p /01_data
RUN mkdir -p /02_code
RUN mkdir -p /03_output
## copy files
COPY /02_code/install_packages.R /02_code/install_packages.R
COPY /02_code/myScript.R /02_code/myScript.R
## install R-packages
RUN Rscript /02_code/install_packages.R
Vergiss nicht, dein entsprechendes install_packages.R-Skript vorzubereiten und zu speichern. Dazu gibst du im Skript an, welche R-Pakete in deinem Image vorinstalliert werden sollen. In unserem Fall sieht die Datei folgendermaßen aus:
install.packages("readr")
install.packages("dplyr")
install.packages("ggplot2")
install.packages("forcats")Erstellen und Ausführen des Images
Nun haben wir alle benötigten Komponenten für unser neues Docker-Image vorbereitet. Verwende das Terminal, um zu dem Ordner zu navigieren, in dem sich dein Dockerfile befindet und erstelle das Image mit:
docker build -t myname/myimage .
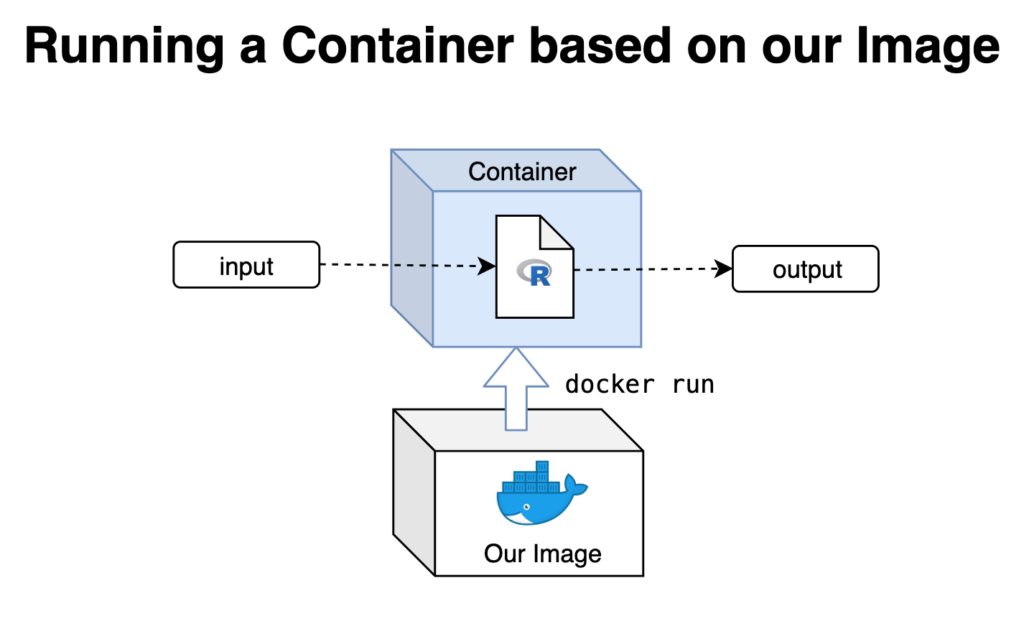
Der Prozess wird aufgrund der Paketinstallation einen Moment dauern. Sobald er abgeschlossen ist, kannst du das neue Image testen, indem du einen Container startest mit:
docker run -it --rm -v ~/"R-Script in Docker"/01_data:/01_data -v ~/"R-Script in Docker"/03_output:/03_output myname/myimage
Die Verwendung der -v-Argumente teilt Docker mit, welche lokalen Ordner den erstellten Ordnern innerhalb des Containers zugeordnet werden sollen. Dies ist wichtig, da wir so sowohl Zugriff auf unseren Datensatz von innerhalb des Containers erhalten, als auch den erstellen Output des Workflows lokal abspeichern können. Dadurch muss der Datensatz nicht in das Image eingebaut werden und wenn das Image gestoppt wird, gehen keine Outputs verloren.
Dieser Container kann nun mit dem Datensatz im Ordner 01_data interagieren und hat eine Kopie unseres Workflow-Skripts in seinem eigenen Ordner 02_code. Wenn Sie R anweisen, source("02_code/myScript.R") auszuführen, wird das Skript ausgeführt und der Output im Ordner 03_output gespeichert, von wo aus er auch in den lokalen Ordner 03_output kopiert wird.
Verbessern was wir haben
Nachdem wir nun getestet und bestätigt haben, dass unser R-Skript im Container wie erwartet läuft, fehlen nur noch ein paar wenige Dinge.
- Wir wollen das Skript nicht manuell aus dem Container heraus aufrufen, sondern es automatisch ausführen lassen, sobald der Container gestartet wird.
Dies können wir ganz einfach erreichen, indem wir den folgenden Befehl an das Ende unserer Dockerdatei anhängen:
## run the script
CMD Rscript /02_code/myScript.R
Das verweist auf den Speicherort unseres Skripts in der Ordnerstruktur des Containers, markiert es als R-Code und weist Docker dann an, beim Starten des Containers das Skript gleich auszuführen. Änderungen am Dockerfile bedeuten natürlich, dass wir unser Image neu erstellen müssen, und das wiederum bedeutet, dass der langsame Prozess der Paketinstallationen wiederholt werden muss. Das ist mühsam, vor allem wenn die Wahrscheinlichkeit besteht, dass es im Laufe der Zeit weitere Überarbeitungen der einzelnen Komponenten unseres Images geben wird. Deshalb schlage ich folgendes vor:
- Erstelle ein Docker-Zwischen-Image, auf dem alle wichtigen Pakete und Abhängigkeiten installiert sind. Auf diesem Zwischen-Image als Basis bauen wir dann unser gewünschtes, finales Image auf.
Auf diese Weise wird die Paketinstallation vom eigentlichen Image entkoppelt, so dass unser finales Image innerhalb von Sekunden neu gebaut werden kann. Dies ermöglicht es uns, frei mit dem Code zu experimentieren, ohne dass wir uns immer wieder mit der Installation von Paketen durch Docker beschäftigen müssen.
Erstellen eines Zwischen-Images
Das Dockerfile für unser Zwischen-Image sieht unserem vorherigen Beispiel sehr ähnlich. Da ich mich entschieden habe, das install_packages()-Skript zu modifizieren, um das gesamte tidyverse für die zukünftige Verwendung einzuschließen, müssen auch einige Debian-Pakete installiert werden, die das tidyverse benötigt. Nicht alle davon sind absolut notwendig, aber alle sind auf die eine oder andere Weise nützlich.
# Base image https://hub.docker.com/u/rocker/
FROM rocker/r-base:latest
# system libraries of general use
## install debian packages
RUN apt-get update -qq && apt-get -y --no-install-recommends install \
libxml2-dev \
libcairo2-dev \
libsqlite3-dev \
libmariadbd-dev \
libpq-dev \
libssh2-1-dev \
unixodbc-dev \
libcurl4-openssl-dev \
libssl-dev
## update system libraries
RUN apt-get update && \
apt-get upgrade -y && \
apt-get clean
## create directories
RUN mkdir -p /02_code
## copy files
COPY /02_code/install_packages.R /02_code/install_packages.R
## install R-packages
RUN Rscript /02_code/install_packages.R
Ich baue das Image, indem ich im Terminal zu dem Ordner, in dem sich mein Dockerfile befindet, navigiere und den Befehl docker build erneut ausführe:
docker build -t oliverstatworx/base-r-tidyverse .
Ich habe dieses Image auch auf meinen DockerHub gepusht. So kannst du, wenn du jemals ein Base-R-Image mit vorinstalliertem tidyverse benötigst, es einfach auf meinem Image aufbauen, ohne dieses selbst erstellen zu müssen.
Nun, da das Zwischen-Image erstellt wurde, können wir unser ursprüngliches Dockerfile so ändern, dass es anstelle von rocker/r-base darauf aufbaut. Da sich unser Zwischen-Image bereits um die Paketinstallation kümmert kann dieser Abschnitt entfernt werden. Wir fügen auch die letzte Zeile hinzu, die unser Skript automatisch ausführt, sobald der Container gestartet wird. Unser endgültiges Dockerfile sollte in etwa so aussehen:
# Base image https://hub.docker.com/u/oliverstatworx/
FROM oliverstatworx/base-r-tidyverse:latest
## create directories
RUN mkdir -p /01_data
RUN mkdir -p /02_code
RUN mkdir -p /03_output
## copy files
COPY /02_code/myScript.R /02_code/myScript.R
## run the script
CMD Rscript /02_code/myScript.RDer letzte Schliff
Da wir unser Image auf einem Zwischen-Image mit all unseren benötigten Paketen aufgebaut haben, können wir nun beliebig Teile des Dockerfiles ohne großen Zeitaufwand verändern. Beispielsweise kann es sinnvoll sein, das R-Skript so zu gestalten, dass Warnungen und Meldungen, die nicht mehr von Interesse sind (da das Image bereits getestet wurde und alles wie erwartet funktioniert) unterdrückt werden. Des weiteren können Meldungen hinzufügt werden, die dem Benutzenden mitteilen, welcher Teil des Skripts gerade von dem laufenden Container ausgeführt wird.
suppressPackageStartupMessages(library(readr))
suppressPackageStartupMessages(library(dplyr))
suppressPackageStartupMessages(library(ggplot2))
suppressPackageStartupMessages(library(forcats))
options(scipen = 999,
readr.num_columns = 0)
print("Starting Workflow")
# import dataframe
print("Importing Dataframe")
df <- read_csv("01_data/us-500.csv")
# manipulate data
print("Manipulating Data")
plot_data <- df %>%
group_by(state) %>%
count()
# save manipulated data to output folder
print("Writing manipulated Data to .csv")
write_csv(plot_data, "03_output/plot_data.csv")
# create plot based on manipulated data
print("Creating Plot")
plot <- plot_data %>%
ggplot()+
geom_col(aes(fct_reorder(state, n),
n,
fill = n))+
coord_flip()+
labs(
title = "Number of people by state",
subtitle = "From US-500 dataset",
x = "State",
y = "Number of people"
)+
theme_bw()
# save plot to output folder
print("Saving Plot")
ggsave("03_output/myplot.png", width = 10, height = 8, dpi = 100)
print("Worflow Finished")
Nachdem wir mit dem Terminal zu dem Ordner navigieren, in dem sich unser Dockerfile befindet, bauen wir das Image noch einmal neu: docker build -t myname/myimage . Erneut starten wir einen Container auf Basis unseres Images und weisen die Ordner 01_data und 03_output den lokalen Verzeichnissen zu, so dass die Daten importiert und die erstellten Outputs lokal gespeichert werden:
docker run -it --rm -v ~/"R-Script in Docker"/01_data:/01_data -v ~/"R-Script in Docker"/03_output:/03_output myname/myimage
Herzlichen Glückwunsch, du hast jetzt ein sauberes Docker-Image erstellt! Dieses führt beim Containerstart nicht nur automatisch dein R-Skript aus, sondern teilt dirauch über Konsolenmeldungen genau mit, welchen Teil des Codes gerade ausgeführt wird. Viel Spaß beim Dockern! Oliver Guggenbühl Oliver Guggenbühl Oliver Guggenbühl Oliver Guggenbühl Oliver Guggenbühl Oliver Guggenbühl


