Wir bei arbeiten viel mit R und verwenden oft die gleichen kleinen Hilfsfunktionen in unseren Projekten. Diese Funktionen erleichtern unseren Arbeitsalltag, indem sie sich-wiederholende Codeteile reduzieren oder Übersichten über unsere Projekte erstellen.
Um diese Funktionen innerhalb unserer Teams und auch mit anderen zu teilen, habe ich angefangen, sie zu sammeln und habe dann daraus ein R-Paket namens erstellt. Neben der gemeinsamen Nutzung wollte ich auch einige Anwendungsfälle haben, um meine Fähigkeiten zur Fehlersuche und Optimierung zu verbessern. Mit der Zeit wuchs das Paket und es kamen immer mehr Funktionen zusammen. Beim letzten Mal habe ich jede Funktion als Teil eines Adventskalenders vorgestellt. Zum Start unserer neuen Website habe ich alle Funktionen in diesem Kalender zusammengefasst und werde jede aktuelle Funktion aus dem Paket vorstellen.
Die meisten Funktionen wurden entwickelt, als es ein Problem gab und man eine einfache Lösung dafür brauchte. Zum Beispiel war der angezeigte Text zu lang und musste gekürzt werden (siehe evenstrings). Andere Funktionen existieren nur, um sich-wiederholende Aufgaben zu reduzieren – wie das Einlesen mehrerer Dateien des selben Typs (siehe read_files). Daher könnten diese Funktionen auch für Euch nützlich sein!
Um alle Funktionen im Detail zu erkunden, könnt Ihr unser besuchen. Wenn Ihr irgendwelche Vorschläge habt, schickt mir bitte eine E-Mail oder öffnet ein Issue auf GitHub!
1. char_replace
Dieser kleine Helfer ersetzt Sonderzeichen (wie z. B. den Umlaut „ä“) durch ihre Standardentsprechung (in diesem Fall „ae“). Es ist auch möglich, alle Zeichen in Kleinbuchstaben umzuwandeln, Leerzeichen zu entfernen oder Leerzeichen und Bindestriche durch Unterstriche zu ersetzen.
Schauen wir uns ein kleines Beispiel mit verschiedenen Settings an:
x <- " Élizàldë-González Strasse"
char_replace(x, to_lower = TRUE)[1] "elizalde-gonzalez strasse"char_replace(x, to_lower = TRUE, to_underscore = TRUE)[1] "elizalde_gonzalez_strasse"char_replace(x, to_lower = FALSE, rm_space = TRUE, rm_dash = TRUE)[1] "ElizaldeGonzalezStrasse"2. checkdir
Dieser kleine Helfer prüft einen gegebenen Ordnerpfad auf Existenz und erstellt ihn bei Bedarf.
checkdir(path = "testfolder/subfolder")Intern gibt es nur eine einfache if-Anweisung, die die R-Basisfunktionen file.exists() und dir.create(). kombiniert.
3. clean_gc
Dieser kleine Helfer gibt den Speicher von unbenutzten Objekten frei. Nun, im Grunde ruft es einfach gc() ein paar Mal auf. Ich habe das vor einiger Zeit für ein Projekt benutzt, bei dem ich mit riesigen Datendateien gearbeitet habe. Obwohl wir das Glück hatten, einen großen Server mit 500 GB RAM zu haben, stießen wir bald an seine Grenzen. Da wir in der Regel mehrere Prozesse parallelisieren, mussten wir jedes Bit und jedes Byte des Arbeitsspeichers nutzen, das wir bekommen konnten. Anstatt also viele Zeilen wie diese zu haben:
gc();gc();gc();gc()… habe ich clean_gc() der Einfachheit halber geschrieben. Intern wird gc() so lange aufgerufen, wie es Speicher gibt, der freigegeben werden muss.
Some further thoughts
Es gibt einige Diskussionen über den Garbage Collector gc() und seine Nützlichkeit. Wenn Ihr mehr darüber erfahren wollt, schlage ich vor, dass Ihr Euch die anseht. Ich weiß, dass R selbst bei Bedarf Speicher freigibt, aber ich bin mir nicht sicher, was passiert, wenn Ihr mehrere R-Prozesse habt. Können sie den Speicher von anderen Prozessen leeren? Wenn Ihr dazu etwas mehr wisst, lasst es mich wissen!
4. count_na
Dieser kleine Helfer zählt fehlende Werte innerhalb eines Vektors.
x <- c(NA, NA, 1, NaN, 0)
count_na(x)3Intern gibt es nur ein einfaches sum(is.na(x)), das die NA-Werte zählt. Wenn Ihr den Mittelwert statt der Summe wollt, könnt Ihr prop = TRUE setzen.
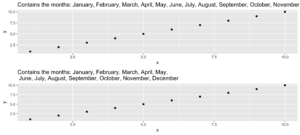
5. evenstrings
Dieser kleine Helfer zerlegt eine gegebene Zeichenkette in kleinere Teile mit einer festen Länge. Aber warum? Nun, ich brauchte diese Funktion beim Erstellen eines Plots mit einem langen Titel. Der Text war zu lang für eine Zeile und anstatt ihn einfach abzuschneiden oder über die Ränder laufen zu lassen, wollte ich ihn schön trennen.
Bei einer langen Zeichenkette wie…
long_title <- c("Contains the months: January, February, March, April, May, June, July, August, September, October, November, December")…wollen wir sie nach split = "," mit einer maximalen Länge von char = 60 aufteilen.
short_title <- evenstrings(long_title, split = ",", char = 60)Die Funktion hat zwei mögliche Ausgabeformate, die durch Setzen von newlines = TRUE oder FALSE gewählt werden können:
- eine Zeichenkette mit Zeilentrennzeichen
\n - ein Vektor mit jedem Unterteil.
Ein anderer Anwendungsfall könnte eine Nachricht sein, die mit cat() auf der Konsole ausgegeben wird:
cat(long_title)Contains the months: January, February, March, April, May, June, July, August, September, October, November, Decembercat(short_title)Contains the months: January, February, March, April, May,
June, July, August, September, October, November, DecemberCode for plot example
p1 <- ggplot(data.frame(x = 1:10, y = 1:10),
aes(x = x, y = y)) +
geom_point() +
ggtitle(long_title)
p2 <- ggplot(data.frame(x = 1:10, y = 1:10),
aes(x = x, y = y)) +
geom_point() +
ggtitle(short_title)
multiplot(p1, p2)6. get_files
Dieser kleine Helfer macht das Gleiche wie die „Find in files „ Suche in RStudio. Sie gibt einen Vektor mit allen Dateien in einem bestimmten Ordner zurück, die das Suchmuster enthalten. In Eurem täglichen Arbeitsablauf würdet Ihr normalerweise die Tastenkombination SHIFT+CTRL+F verwenden. Mit get_files() könnt Ihr diese Funktionen in Euren Skripten nutzen.
7. get_network
Das Ziel dieses kleinen Helfers ist es, die Verbindungen zwischen R-Funktionen innerhalb eines Projekts als Flussdiagramm zu visualisieren. Dazu ist die Eingabe ein Verzeichnispfad zur Funktion oder eine Liste mit den Funktionen und die Ausgaben sind eine Adjazenzmatrix und ein Graph-Objekt. Als Beispiel verwenden wir diesen Ordner mit einigen Spielzeugfunktionen:
net <- get_network(dir = "flowchart/R_network_functions/", simplify = FALSE)
g1 <- net$igraphInput
Es gibt fünf Parameter, um mit der Funktion zu interagieren:
- ein Pfad
dir, der durchsucht werden soll. - ein Zeichenvektor
Variationenmit der Definitionszeichenfolge der Funktion – die Vorgabe istc(" <- function", "<- function", "<-function"). - ein „Muster“, eine Zeichenkette mit dem Dateisuffix – die Vorgabe ist
"\\.R$". - ein boolesches
simplify, das Funktionen ohne Verbindungen aus der Darstellung entfernt. - eine benannte Liste
all_scripts, die eine Alternative zudirist. Diese Liste wird hauptsächlich nur zu Testzwecken verwendet.
Für eine normale Verwendung sollte es ausreichen, einen Pfad zum Projektordner anzugeben.
Output
Der gegebene Plot zeigt die Verbindungen der einzelnen Funktionen (Pfeile) und auch die relative Größe des Funktionscodes (Größe der Punkte). Wie bereits erwähnt, besteht die Ausgabe aus einer Adjazenzmatrix und einem Graph-Objekt. Die Matrix enthält die Anzahl der Aufrufe für jede Funktion. Das Graph-Objekt hat die folgenden Eigenschaften:
- Die Namen der Funktionen werden als Label verwendet.
- Die Anzahl der Zeilen jeder Funktion (ohne Kommentare und Leerzeilen) wird als Größe gespeichert.
- Der Ordnername des ersten Ordners im Verzeichnis.
- Eine Farbe, die dem Ordner entspricht.
Mit diesen Eigenschaften können Sie die Netzwerkdarstellung zum Beispiel wie folgt verbessern:
library(igraph)
# create plots ------------------------------------------------------------
l <- layout_with_fr(g1)
colrs <- rainbow(length(unique(V(g1)$color)))
plot(g1,
edge.arrow.size = .1,
edge.width = 5*E(g1)$weight/max(E(g1)$weight),
vertex.shape = "none",
vertex.label.color = colrs[V(g1)$color],
vertex.label.color = "black",
vertex.size = 20,
vertex.color = colrs[V(g1)$color],
edge.color = "steelblue1",
layout = l)
legend(x = 0,
unique(V(g1)$folder), pch = 21,
pt.bg = colrs[unique(V(g1)$color)],
pt.cex = 2, cex = .8, bty = "n", ncol = 1)
Dieser kleine Helfer gibt Indizes von wiederkehrenden Mustern zurück. Es funktioniert sowohl mit Zahlen als auch mit Zeichen. Alles, was es braucht, ist ein Vektor mit den Daten, ein Muster, nach dem gesucht werden soll, und eine Mindestanzahl von Vorkommen.
Lasst uns mit dem folgenden Code einige Zeitreihendaten erstellen.
library(data.table)
# random seed
set.seed(20181221)
# number of observations
n <- 100
# simulationg the data
ts_data <- data.table(DAY = 1:n, CHANGE = sample(c(-1, 0, 1), n, replace = TRUE))
ts_data[, VALUE := cumsum(CHANGE)]Dies ist nichts anderes als ein Random Walk, da wir zwischen dem Abstieg (-1), dem Anstieg (1) und dem Verbleib auf demselben Niveau (0) wählen. Unsere Zeitreihendaten sehen folgendermaßen aus:
Angenommen, wir wollen die Datumsbereiche wissen, in denen es an mindestens vier aufeinanderfolgenden Tagen keine Veränderung gab.
ts_data[, get_sequence(x = CHANGE, pattern = 0, minsize = 4)] min max
[1,] 45 48
[2,] 65 69Wir können auch die Frage beantworten, ob sich das Muster „down-up-down-up“ irgendwo wiederholt:
ts_data[, get_sequence(x = CHANGE, pattern = c(-1,1), minsize = 2)] min max
[1,] 88 91Mit diesen beiden Eingaben können wir unseren Plot ein wenig aktualisieren, indem wir etwas geom_rect hinzufügen!
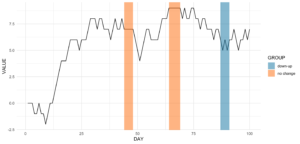
Code for the plot
rect <- data.table(
rbind(ts_data[, get_sequence(x = CHANGE, pattern = c(0), minsize = 4)],
ts_data[, get_sequence(x = CHANGE, pattern = c(-1,1), minsize = 2)]),
GROUP = c("no change","no change","down-up"))
ggplot(ts_data, aes(x = DAY, y = VALUE)) +
geom_line() +
geom_rect(data = rect,
inherit.aes = FALSE,
aes(xmin = min - 1,
xmax = max,
ymin = -Inf,
ymax = Inf,
group = GROUP,
fill = GROUP),
color = "transparent",
alpha = 0.5) +
scale_fill_manual(values = statworx_palette(number = 2, basecolors = c(2,5))) +
theme_minimal()9. intersect2
Dieser kleine Helfer gibt den Schnittpunkt mehrerer Vektoren oder Listen zurück. Ich habe diese Funktion gefunden, fand sie recht nützlich und habe sie ein wenig angepasst.
intersect2(list(c(1:3), c(1:4)), list(c(1:2),c(1:3)), c(1:2))[1] 1 2Intern wird das Problem, die Schnittmenge zu finden, rekursiv gelöst, wenn ein Element eine Liste ist, und dann schrittweise mit dem nächsten Element.
10. multiplot
Dieses kleine Hilfsmittel kombiniert mehrere ggplots zu einem Plot. Dies ist eine Funktion aus .
Ein Vorteil gegenüber facets ist, dass man nicht alle Daten für alle Plots in einem Objekt benötigt. Auch kann man jeden einzelnen Plot frei erstellen – was manchmal auch ein Nachteil sein kann.
Mit dem Parameter layout könnt Ihr mehrere Plots mit unterschiedlichen Größen anordnen. Nehmen wir an, Ihr habt drei Plots und wollt sie wie folgt anordnen:
1 2 2
1 2 2
3 3 3Bei multiplot läuft es auf Folgendes hinaus:
multiplot(plotlist = list(p1, p2, p3),
layout = matrix(c(1,2,2,1,2,2,3,3,3), nrow = 3, byrow = TRUE))Code for plot example
# star coordinates
c1 = cos((2*pi)/5)
c2 = cos(pi/5)
s1 = sin((2*pi)/5)
s2 = sin((4*pi)/5)
data_star <- data.table(X = c(0, -s2, s1, -s1, s2),
Y = c(1, -c2, c1, c1, -c2))
p1 <- ggplot(data_star, aes(x = X, y = Y)) +
geom_polygon(fill = "gold") +
theme_void()
# tree
set.seed(24122018)
n <- 10000
lambda <- 2
data_tree <- data.table(X = c(rpois(n, lambda), rpois(n, 1.1*lambda)),
TYPE = rep(c("1", "2"), each = n))
data_tree <- data_tree[, list(COUNT = .N), by = c("TYPE", "X")]
data_tree[TYPE == "1", COUNT := -COUNT]
p2 <- ggplot(data_tree, aes(x = X, y = COUNT, fill = TYPE)) +
geom_bar(stat = "identity") +
scale_fill_manual(values = c("green", "darkgreen")) +
coord_flip() +
theme_minimal()
# gifts
data_gifts <- data.table(X = runif(5, min = 0, max = 10),
Y = runif(5, max = 0.5),
Z = sample(letters[1:5], 5, replace = FALSE))
p3 <- ggplot(data_gifts, aes(x = X, y = Y)) +
geom_point(aes(color = Z), pch = 15, size = 10) +
scale_color_brewer(palette = "Reds") +
geom_point(pch = 12, size = 10, color = "gold") +
xlim(0,8) +
ylim(0.1,0.5) +
theme_minimal() +
theme(legend.position="none")
11. na_omitlist
Dieser kleine Helfer entfernt fehlende Werte aus einer Liste.
y <- list(NA, c(1, NA), list(c(5:6, NA), NA, "A"))Es gibt zwei Möglichkeiten, die fehlenden Werte zu entfernen, entweder nur auf der ersten Ebene der Liste oder innerhalb jeder Unterebene.
na_omitlist(y, recursive = FALSE)[[1]]
[1] 1 NA
[[2]]
[[2]][[1]]
[1] 5 6 NA
[[2]][[2]]
[1] NA
[[2]][[3]]
[1] "A"na_omitlist(y, recursive = TRUE)[[1]]
[1] 1
[[2]]
[[2]][[1]]
[1] 5 6
[[2]][[2]]
[1] "A"12. %nin%
Dieser kleine Helfer ist eine reine Komfortfunktion. Sie ist einfach dasselbe wie der negierte %in%-Operator, wie Ihr unten sehen könnt. Aber meiner Meinung nach erhöht sie die Lesbarkeit des Codes.
all.equal( c(1,2,3,4) %nin% c(1,2,5),
!c(1,2,3,4) %in% c(1,2,5))[1] TRUEDieser Operator hat es auch in einige andere Pakete geschafft – .
13. object_size_in_env
Dieser kleine Helfer zeigt eine Tabelle mit der Größe jedes Objekts in der vorgegebenen Umgebung an.
Wenn Ihr in einer Situation seid, in der Ihr viel gecodet habt und Eure Umgebung nun ziemlich unübersichtlich ist, hilft Euch object_size_in_env, die großen Fische in Bezug auf den Speicherverbrauch zu finden. Ich selbst bin ein paar Mal auf dieses Problem gestoßen, als ich mehrere Ausführungen meiner Modelle in einem Loop durchlaufen habe. Irgendwann wurden die Sitzungen ziemlich groß im Speicher und ich wusste nicht, warum! Mit Hilfe von object_size_in_env und etwas Degubbing konnte ich das Objekt ausfindig machen, das dieses Problem verursachte, und meinen Code entsprechend anpassen.
Zuerst wollen wir eine Umgebung mit einigen Variablen erstellen.
# building an environment
this_env <- new.env()
assign("Var1", 3, envir = this_env)
assign("Var2", 1:1000, envir = this_env)
assign("Var3", rep("test", 1000), envir = this_env)Um die Größeninformationen unserer Objekte zu erhalten, wird intern format(object.size()) verwendet. Mit der Einheit kann das Ausgabeformat geändert werden (z.B. "B", "MB" oder "GB").
# checking the size
object_size_in_env(env = this_env, unit = "B") OBJECT SIZE UNIT
1: Var3 8104 B
2: Var2 4048 B
3: Var1 56 B14. print_fs
Dieser kleine Helfer gibt die Ordnerstruktur eines gegebenen Pfades zurück. Damit kann man z.B. eine schöne Übersicht in die Dokumentation eines Projektes oder in ein Git einbauen. Im Sinne der Automatisierung könnte diese Funktion nach einer größeren Änderung Teile in einer Log- oder News-Datei ändern.
Wenn wir uns das gleiche Beispiel anschauen, das wir für die Funktion get_network verwendet haben, erhalten wir folgendes:
print_fs("~/flowchart/", depth = 4)1 flowchart
2 ¦--create_network.R
3 ¦--getnetwork.R
4 ¦--plots
5 ¦ ¦--example-network-helfRlein.png
6 ¦ °--improved-network.png
7 ¦--R_network_functions
8 ¦ ¦--dataprep
9 ¦ ¦ °--foo_01.R
10 ¦ ¦--method
11 ¦ ¦ °--foo_02.R
12 ¦ ¦--script_01.R
13 ¦ °--script_02.R
14 °--README.md Mit depth können wir einstellen, wie tief wir unsere Ordner durchforsten wollen.
15. read_files
Dieser kleine Helfer liest mehrere Dateien des selben Typs ein und fasst sie zu einer data.table zusammen. Welche Art von Dateilesefunktion verwendet werden soll, kann mit dem Argument FUN ausgewählt werden.
Wenn Sie eine Liste von Dateien haben, die alle mit der gleichen Funktion eingelesen werden sollen (z.B. read.csv), können Sie statt lapply und rbindlist nun dies verwenden:
read_files(files, FUN = readRDS)
read_files(files, FUN = readLines)
read_files(files, FUN = read.csv, sep = ";")Intern verwendet es nur lapply und rbindlist, aber man muss es nicht ständig eingeben. Die read_files kombiniert die einzelnen Dateien nach ihren Spaltennamen und gibt eine data.table zurück. Warum data.table? Weil ich es mag. Aber lassen Sie uns nicht das Fass von data.table vs. dplyr aufmachen (zum Fass…).
16. save_rds_archive
Dieser kleine Helfer ist ein Wrapper um die Basis-R-Funktion saveRDS() und prüft, ob die Datei, die Ihr zu speichern versucht, bereits existiert. Wenn ja, wird die bestehende Datei umbenannt / archiviert (mit einem Zeitstempel), und die „aktualisierte“ Datei wird unter dem angegebenen Namen gespeichert. Das bedeutet, dass vorhandener Code, der davon abhängt, dass der Dateiname konstant bleibt (z.B. readRDS()-Aufrufe in anderen Skripten), weiterhin funktionieren wird, während eine archivierte Kopie der – ansonsten überschriebenen – Datei erhalten bleibt.
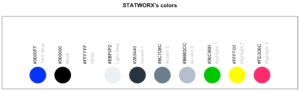
17. sci_palette
Dieser kleine Helfer liefert eine Reihe von Farben, die wir bei statworx häufig verwenden. Wenn Ihr Euch also – so wie ich – nicht an jeden Hex-Farbcode erinnern könnt, den Ihr braucht, könnte das helfen. Natürlich sind das unsere Farben, aber Ihr könnt es auch mit Eurer eigenen Farbpalette umschreiben. Aber der Hauptvorteil ist die Plot-Methode – so könnt Ihr die Farbe sehen, anstatt nur den Hex-Code zu lesen.
So seht Ihr, welcher Hexadezimalcode welcher Farbe entspricht und wofür Ihr ihn verwenden könnt.
sci_palette(scheme = "new")Tech Blue Black White Light Grey Accent 1 Accent 2 Accent 3
"#0000FF" "#000000" "#FFFFFF" "#EBF0F2" "#283440" "#6C7D8C" "#B6BDCC"
Highlight 1 Highlight 2 Highlight 3
"#00C800" "#FFFF00" "#FE0D6C"
attr(,"class")
[1] "sci"Wie bereits erwähnt, gibt es eine Methode plot(), die das folgende Bild ergibt.
plot(sci_palette(scheme = "new"))
18. statusbar
Dieser kleine Helfer gibt einen Fortschrittsbalken in der Konsole für Schleifen aus.
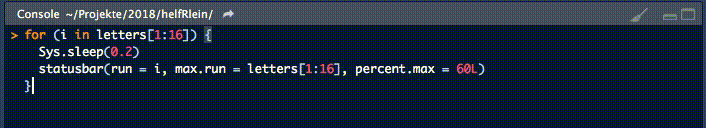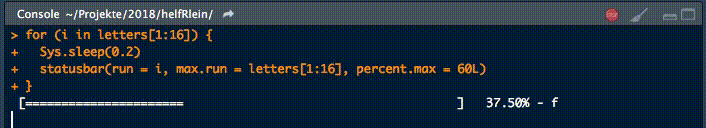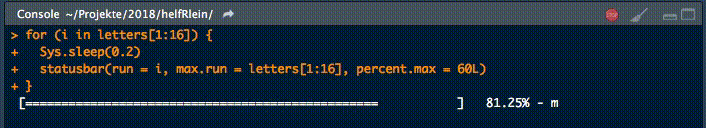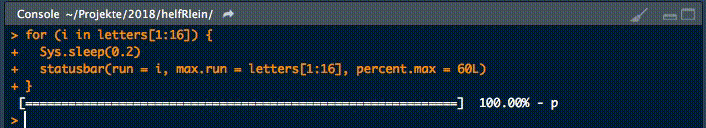
Es gibt zwei notwendige Parameter, um diese Funktion zu füttern:
runist entweder der Iterator oder seine Nummermax.runist entweder alle möglichen Iteratoren in der Reihenfolge, in der sie verarbeitet werden, oder die maximale Anzahl von Iterationen.
So könnte es zum Beispiel run = 3 und max.run = 16 oder run = "a" und max.run = Buchstaben[1:16] sein.
Außerdem gibt es zwei optionale Parameter:
percent.maxbeeinflusst die Breite des Fortschrittsbalkensinfoist ein zusätzliches Zeichen, das am Ende der Zeile ausgegeben wird. Standardmäßig ist esrun.
Ein kleiner Nachteil dieser Funktion ist, dass sie nicht mit parallelen Prozessen arbeitet. Wenn Ihr einen Fortschrittsbalken haben wollt, wenn Ihr apply Funktionen benutzt, schaut Euch an.
19. statworx_palette
Dieses kleine Hilfsmittel ist eine Ergänzung zu sci_palette(). Wir haben die Farben 1, 2, 3, 5 und 10 ausgewählt, um eine flexible Farbpalette zu erstellen. Wenn Sie 100 verschiedene Farben benötigen – sagen Sie nichts mehr!
Im Gegensatz zu sci_palette() ist der Rückgabewert ein Zeichenvektor. Zum Beispiel, wenn Sie 16 Farben wollen:
statworx_palette(16, scheme = "old")[1] "#013848" "#004C63" "#00617E" "#00759A" "#0087AB" "#008F9C" "#00978E" "#009F7F"
[9] "#219E68" "#659448" "#A98B28" "#ED8208" "#F36F0F" "#E45A23" "#D54437" "#C62F4B"Wenn wir nun diese Farben aufzeichnen, erhalten wir einen schönen regenbogenartigen Farbverlauf.
library(ggplot2)
ggplot(plot_data, aes(x = X, y = Y)) +
geom_point(pch = 16, size = 15, color = statworx_palette(16, scheme = "old")) +
theme_minimal()
Eine zusätzliche Funktion ist der Parameter reorder, der die Reihenfolge der Farben abtastet, so dass Nachbarn vielleicht etwas besser unterscheidbar sind. Auch wenn Sie die verwendeten Farben ändern wollen, können Sie dies mit basecolors tun.
ggplot(plot_data, aes(x = X, y = Y)) +
geom_point(pch = 16, size = 15,
color = statworx_palette(16, basecolors = c(4,8,10), scheme = "new")) +
theme_minimal()
20. strsplit
Dieses kleine Hilfsmittel erweitert die R-Basisfunktion strsplit – daher der gleiche Name! Es ist nun möglich, before, after oder between ein bestimmtes Begrenzungszeichen zu trennen. Im Falle von between müsst ihr zwei Delimiter angeben.
Eine frühere Version dieser Funktion findet Ihr in diesem Blogbeitrag, wo ich die verwendeten regulären Ausdrücke beschreibe, falls Ihr daran interessiert seid.
Hier ist ein kleines Beispiel, wie man das neue strsplit benutzt.
text <- c("This sentence should be split between should and be.")
strsplit(x = text, split = " ")
strsplit(x = text, split = c("should", " be"), type = "between")
strsplit(x = text, split = "be", type = "before")[[1]]
[1] "This" "sentence" "should" "be" "split" "between" "should" "and"
[9] "be."
[[1]]
[1] "This sentence should" " be split between should and be."
[[1]]
[1] "This sentence should " "be split " "between should and "
[4] "be."21. to_na
Dieser kleine Helfer ist nur eine Komfortfunktion. Bei der Datenaufbereitung kann es vorkommen, dass Ihr einen Vektor mit unendlichen Werten wie Inf oder -Inf oder sogar NaN-Werten habt. Solche Werte können (müssen aber nicht!) Eure Auswertungen und Modelle durcheinanderbringen. Aber die meisten Funktionen haben die Tendenz, fehlende Werte zu behandeln. Daher entfernt diese kleine Hilfe solche Werte und ersetzt sie durch NA.
Ein kleines Beispiel, um Euch die Idee zu vermitteln:
test <- list(a = c("a", "b", NA),
b = c(NaN, 1,2, -Inf),
c = c(TRUE, FALSE, NaN, Inf))
lapply(test, to_na)$a
[1] "a" "b" NA
$b
[1] NA 1 2 NA
$c
[1] TRUE FALSE NAEin kleiner Tipp am Rande! Da es je nach den anderen Werten innerhalb eines Vektors verschiedene Arten von NA gibt, solltet Ihr das Format überprüfen, wenn Ihr to_na auf Gruppen oder Teilmengen anwendet.
test <- list(NA, c(NA, "a"), c(NA, 2.3), c(NA, 1L))
str(test)List of 4
$ : logi NA
$ : chr [1:2] NA "a"
$ : num [1:2] NA 2.3
$ : int [1:2] NA 122. trim
Dieser kleine Helfer entfernt führende und nachfolgende Leerzeichen aus einer Zeichenkette. Mit wurde trimws eingeführt, das genau das Gleiche tut. Das zeigt nur, dass es keine schlechte Idee war, eine solche Funktion zu schreiben. 😉
x <- c(" Hello world!", " Hello world! ", "Hello world! ")
trim(x, lead = TRUE, trail = TRUE)[1] "Hello world!" "Hello world!" "Hello world!"Die Parameter lead und trail geben an, ob nur die führenden, die nachfolgenden oder beide Leerzeichen entfernt werden sollen.
Fazit
Ich hoffe, dass euch das helfRlein Package genauso die Arbeit erleichtert, wie uns hier bei statworx. Schreibt uns bei Fragen oder Input zum Package gerne eine Mail an: blog@statworx.com
Im Bereich Data Science – wie der Name schon sagt – ist das Thema Daten, vom Data Cleaning bis hin zum Feature Engineering, einer der Grundpfeiler. Daten zu haben und auszuwerten ist die eine Seite, doch wie kommt man eigentlich an Daten für neue Problemstellungen?
Wenn man Glück hat, werden die Daten, die man benötigt, bereits zur Verfügung gestellt. Sei es über den Download eines ganzen Datensatzes oder die Verwendung einer API. Häufig muss man allerdings auch Informationen von Webseiten selbst zusammentragen – das nennt man Web Scraping. Je nachdem wie oft man Daten scrapen will, ist es von Vorteil, diesen Schritt zu automatisieren.
In diesem Beitrag soll es genau um diese Automatisierung gehen. Ich werde mittels Web Scraping und GitHub Actions an einem Beispiel aufzeigen, wie man sich selbst Datensätze über einen längeren Zeitraum erstellen kann. Dabei soll der Fokus auf den Erfahrungen liegen, die ich in den letzten Monaten gesammelt habe.
Der verwendete Code sowie die bisher gesammelten Daten befinden sich in diesem GitHub Repo.
Suche nach Daten – Ausgangslage
Bei meiner Recherche für den Blogbeitrag über die Benzinpreise, bin ich auch über Daten zur Auslastung der Parkhäuser in Frankfurt am Main gestoßen. Die Beschaffung dieser Daten legte den Grundstein für diesen Beitrag. Nach einigen Überlegungen und zusätzlicher Recherche kamen mir noch weitere thematisch passende Datenquellen in den Sinn:
- Auslastung der Straßen
- Verspätungen der S- und U-Bahnen
- Events in der Nähe
- Wetterdaten
Schnell stellte sich jedoch heraus, dass ich nicht alle diese Daten bekommen konnte, da sie nicht frei verfügbar sind bzw. es nicht gestattet ist, diese zu speichern. Da ich vorhatte, die gesammelten Daten auf GitHub zu speichern und verfügbar zu machen, war dies ein entscheidender Punkt, welche Daten in Frage kamen. Aus diesen Gründen fielen die Bahndaten vollkommen raus. Für die Straßenauslastung habe ich lediglich Daten für Köln gefunden und ich wollte es vermeiden, die Google API zu nutzen, da das durchaus seine eigenen Herausforderungen mit sich bringt. Es blieben also Event- und Wetterdaten.
Für die Wetterdaten des Deutschen Wetterdienstes kann das rdwd Packet genutzt werden. Da diese Daten bereits historisiert vorliegen, sind sie für diesen Blogbeitrag nebensächlich. Um an die verbleibenden Event- und Parkdaten zu kommen, haben sich die GitHub Actions als sehr nützlich erwiesen – auch wenn sie nicht ganz trivial in der Anwendung sind. Besonders der Umstand, dass diese kostenfrei genutzt werden können, machen sie zu einem empfehlenswerten Tool für solche Projekte.
Scrapen der Daten
Da sich dieser Beitrag nicht mit Details zum Thema Webscraping befassen wird, verweise ich an dieser Stelle auf den Beitrag von meinem Kollegen David.
Die Parkdaten stehen hier im XML-Format bereit und werden alle fünf Minuten aktualisiert. Sobald man die Struktur des XML verstanden hat, müsst ihr nur noch auf den richtigen Index zugreifen und ihr habt die Daten, die ihr möchtet.
In der Funktion get_parking_data() habe ich alles zusammengefasst, was ich benötige. Es wird ein Datensatz zur Area und ein Datensatz zu den einzelnen Parkhäusern erstellt.
Beispiel Datenauszug area
parkingAreaOccupancy;parkingAreaStatusTime;parkingAreaTotalNumberOfVacantParkingSpaces;
totalParkingCapacityLongTermOverride;totalParkingCapacityShortTermOverride;id;TIME
0.08401977;2021-12-01T01:07:00Z;556;150;607;1[Anlagenring];2021-12-01T01:07:02.720Z
0.31417114;2021-12-01T01:07:00Z;513;0;748;4[Bahnhofsviertel];2021-12-01T01:07:02.720Z
0.351417;2021-12-01T01:07:00Z;801;0;1235;5[Dom / Römer];2021-12-01T01:07:02.720Z
0.21266666;2021-12-01T01:07:00Z;1181;70;1500;2[Zeil];2021-12-01T01:07:02.720Z
Beispiel Datenauszug facility
parkingFacilityOccupancy;parkingFacilityStatus;parkingFacilityStatusTime;
totalNumberOfOccupiedParkingSpaces;totalNumberOfVacantParkingSpaces;
totalParkingCapacityLongTermOverride;totalParkingCapacityOverride;
totalParkingCapacityShortTermOverride;id;TIME
0.02;open;2021-12-01T01:02:00Z;4;196;150;350;200;24276[Turmcenter];2021-12-01T01:07:02.720Z
0.11547912;open;2021-12-01T01:02:00Z;47;360;0;407;407;18944[Alte Oper];2021-12-01T01:07:02.720Z
0.0027472528;open;2021-12-01T01:02:00Z;1;363;0;364;364;24281[Hauptbahnhof Süd];2021-12-01T01:07:02.720Z
0.609375;open;2021-12-01T01:02:00Z;234;150;0;384;384;105479[Baseler Platz];2021-12-01T01:07:02.720Z
Für die Eventdaten scrape ich die Seite stadtleben.de. Da es sich um eine HTML handelt, die recht gut strukturiert ist, kann ich über den Tag „kalenderListe“ auf die tabellarische Eventübersicht zugreifen. Das Resultat wird durch die Funktion get_event_data() erstellt.
Beispiel Datenauszug events
eventtitle;views;place;address;eventday;eventdate;request
Magical Sing Along - Das lustigste Mitsing-Event;12576;Bürgerhaus;64546 Mörfelden-Walldorf, Westendstraße 60;Freitag;2022-03-04;2022-03-04T02:24:14.234833Z
Velvet-Bar-Night;1460;Velvet Club;60311 Frankfurt, Weißfrauenstraße 12-16;Freitag;2022-03-04;2022-03-04T02:24:14.234833Z
Basta A-cappella-Band;465;Zeltpalast am Deutsche Bank Park;60528 Frankfurt am Main, Mörfelder Landstraße 362;Freitag;2022-03-04;2022-03-04T02:24:14.234833Z
BeThrifty Vintage Kilo Sale | Frankfurt | 04. & 05. …;1302;Batschkapp;60388 Frankfurt am Main, Gwinnerstraße 5;Freitag;2022-03-04;2022-03-04T02:24:14.234833Z
Automation der Abläufe – GitHub Actions
Das Grundgerüst steht. Ich habe je eine Funktion, die mir die Park- und Eventdaten beim Ausführen in eine .csv Datei schreiben. Da ich die Parkdaten alle fünf Minuten und die Eventdaten zur Sicherheit drei Mal am Tag abfragen möchte, kommen nun GitHub Actions ins Spiel.
Mit dieser Funktion von GitHub können neben Aktionen, die beim Mergen oder Committen auslösen, auch Workflows zeitlich geplant und durchgeführt werden. Hierfür wird eine .yml Datei im Order /.github/workflows erstellt.
Die Hauptbestandteile meines Workflows sind:
- Der
schedule– Alle zehn Minuten sollen die Funktionen ausgeführt werden. - Das OS – Da ich lokal auf einem Mac entwickle, nutze ich hier das
macOS-latest. - Umgebungsvariablen – Hier ist neben meinem GitHub Token auch der Pfad für das Paketmanagement
renventhalten - Die einzelnen
stepsim Workflow selbst
Der Workflow durchläuft die folgenden Schritte:
- Setup R
- Pakete laden mit renv
- Script ausführen um Daten zu scrapen
- Script ausführen um die README zu aktualisieren
- Pushen der neuen Daten zurück ins git
Jeder dieser Schritte ist an sich sehr klein und übersichtlich, jedoch liegt der Teufel wie so oft im Detail.
Limitation und Herausforderungen
Im Laufe der letzten Monate habe ich meinen Workflow immer wieder angepasst und optimiert, um den aufkommenden Fehlern und Problemen Herr zu werden. Nachfolgend also der Überblick über meine kondensierten Erfahrungen mit GitHub Actions.
Schedule Probleme
Wer zeitkritische Aktionen durchführen möchte, sollte auf andere Services zugreifen. GitHub Actions garantieren einem nicht, dass die Jobs exakt getimed werden (oder teilweise überhaupt durchgeführt werden). In der Tabelle sind die Zeiten zwischen zwei erfolgreichen Abfragen angegeben.
| Zeitspanne in Minuten | <= 5 | <= 10 | <= 20 | <= 60 | > 60 |
| Anzahl Abfragen | 1720 | 2049 | 5509 | 3023 | 194 |
Man sieht, dass die geplanten fünf Minuten Intervalle nicht immer eingehalten wurden. Hier sollte ich in Zukunft einen größeren Spielraum einplanen.
Merge Konflikte
Zu Beginn hatte ich zwei Workflows, einen für die Parkdaten und einen für die Events. Wenn diese sich zeitlich überlappt haben, dann kam es zu Merge-Konflikten, da beide Prozesse die README mit einen Zeitstempel updaten. Im Verlauf bin ich umgestiegen auf einen Workflow samt Errorhandling.
Auch wenn ein Durchlauf länger gedauert hat und der nächste bereits gestartet wurde, kam es beim Pushen zu Merge-Konflikten in den .csv-Daten. Lange Durchläufe entstanden häufig durch das R Setup und das Laden der packages. Als Konsequenz habe ich das Schedule-Intervall von fünf auf zehn Minuten erweitert.
Formatanpassungen
Es gab ein paar Situationen, in denen sich die Pfade oder Struktur der gescrapten Daten geändert haben, so dass ich meine Funktionen anpassen musste. Hierbei war die Einstellung, eine E-Mail zu bekommen, falls ein Prozess gescheitert ist, sehr hilfreich.
Fehlende Testmöglichkeiten
Es gibt bisher keine andere Möglichkeit ein Workflow-Script zu testen, als es wirklich laufen zu lassen. So kann man nach einem Tippfehler am Abend zu einer Mailflut mit gefailten Runs am Morgen aufwachen. Das sollte einen dennoch nicht davon abhalten einen lokalen Testlauf durchzuführen.
Kein Datenupdate
Seit Ende Dezember wurden die Parkdaten nicht mehr aktualisiert bzw. bereitgestellt. Das zeigt, dass selbst wenn man einen automatischen Prozess hat, man ihn dennoch weiter überwachen sollte. Ich habe dies erst später festgestellt, wodurch meine Abfragen Ende Dezember immer ins Leere liefen.
Fazit
Trotz der Komplikationen aus dem letzten Kapitel, empfinde ich das Ganze dennoch als einen massiven Erfolg. Während der letzten Monate habe ich mich immer wieder mit dem Thema befasst und die oben beschriebenen Tricks und Kniffe erlernt, die mir auch in Zukunft helfen werden, andere Probleme zu lösen. Ich hoffe, dass auch ihr ein paar wertvolle Hinweise mitnehmen und somit aus meinen Fehlern lernen könnt.
Da ich nun ein gutes halbes Jahr an Daten gesammelt haben, kann ich mich mit der Auswertung befassen. Das wird dann aber erst Gegenstand eines weiteren Blogbeitrages.
In meinem ersten Blogbeitrag dieser Reihe habe ich gezeigt, wie du deine R-Skripte in einem Docker-Container ausführen kannst. Für viele der Projekte, an denen wir hier bei STATWORX arbeiten, verwenden wir das RShiny-Framework, um unsere Produkte in interaktive Webapplikationen zu verwandeln.
Die Verwendung von Containern hat für die Bereitstellung von ShinyApps eine Vielzahl von Vorteilen. Zunächst sind es die üblichen Vorzüge wie einfache Cloud-Bereitstellung, Skalierbarkeit und praktisches Scheduling, aber es behebt auch einen der wesentlichen Nachteile von Shiny: Shiny erstellt nur eine einzige R-Sitzung pro App. Sollten also mehrere User auf dieselbe App zugreifen, dann arbeiten sie alle mit der selben R-Sitzung, was zu einer Vielzahl von Problemen führt. Mithilfe von Docker können wir dieses Problem umgehen und für jede:n User:in eine eigene Container-Instanz starten. Dadurch erhält jede:r User:in Zugriff auf eine eigene Instanz der App und somit eine eigene R-Sitzung. Ich gehe davon aus, dass du meinen vorigen Blogbeitrag über das Einbinden von R-Skripten in ein Docker-Image gelesen hast oder bereits über Grundkenntnisse der Docker-Terminologie verfügst.
Lassen wir also einfache R-Skripte hinter uns und führen wir jetzt ganze ShinyApps in Docker aus!
Das Setup
Einrichten eines Projekts
Für die Arbeit mit ShinyApps ist es ratsam, das Projekt-Setup von RStudio zu nutzen, insbesondere wenn man Docker verwendet. Projekte erleichtern nicht nur die Ordnung in RStudio, sondern ermöglichen es uns auch, mit dem Paket renv eine Paketbibliothek für unser spezifisches Projekt einzurichten. Dies ist besonders praktisch, um die benötigten Pakete für ein Programm in ein Docker-Image zu installieren.
Zu Demonstrationszwecken verwende ich eine Beispiel-App, die in einem früheren Blogbeitrag erstellt wurde und die du aus dem STATWORX GitHub Repository klonen kannst. Sie befindet sich im Unterordner „example-app“ und besteht aus den drei typischen Skripten, die von ShinyApps genutzt werden (global.R, ui.R und server.R) sowie aus Dateien, die zur Paketbibliothek renv gehören. Solltest du zur Übung ebenfalls die oben verlinkte Beispiel-App verwenden, musst du kein eigenes RStudio-Projekt einrichten. Stattdessen kannst du example-app.Rproj öffnen, das den von mir bereits eingerichteten Projektkontext startet. Falls du direkt mit einer eigenen App arbeiten möchtest und noch kein Projekt dafür erstellt hast, kannst du dein eigenes Projekt einrichten, indem du die von RStudio bereitgestellten Anweisungen befolgst.
Einrichten einer Paketbibliothek
Das RStudio-Projekt, das ich zur Verfügung gestellt habe, wird bereits mit einer Paketbibliothek geliefert, die in der Datei renv.lock gespeichert ist. Wenn du es vorziehst, mit deiner eigenen Anwendung zu arbeiten, kannst du deine eigene renv.lock Datei erstellen, indem du das renv Paket von deinem RStudio Projekt aus installierst und renv::init() ausführst. Dies initialisiert renv für dein Projekt und erstellt eine renv.lock Datei in deinem Projekt-Stammverzeichnis. Mehr Informationen über renv findest du unter RStudio’s Einführungsartikel.
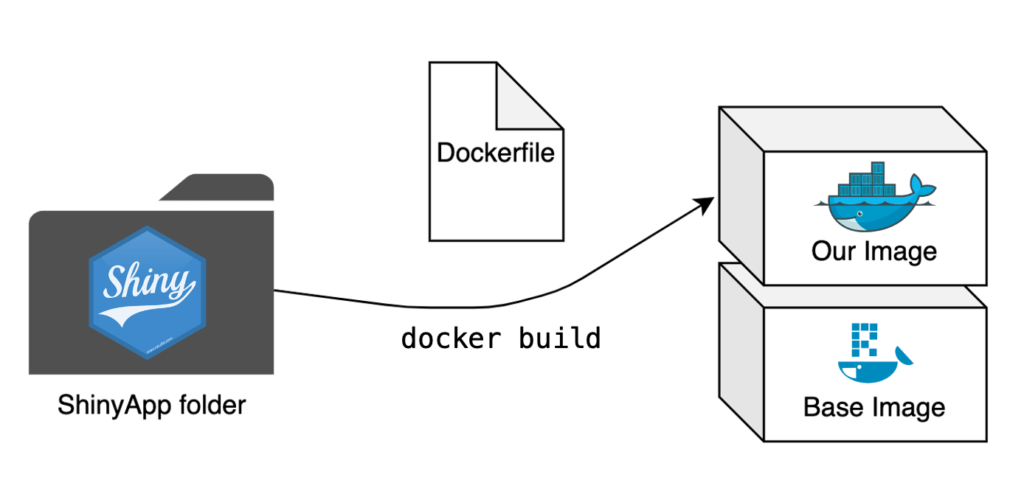
Das Dockerfile
Das Dockerfile ist erneut das zentrale Element bei der Erstellung des Docker-Images. Während wir bisher nur ein einziges Skript in ein Image eingebaut haben, wollen wir diesen Prozess nun für eine ganze Anwendung wiederholen. Der Schritt von einem einzelnen Skript zu einem Ordner mit mehreren Skripten ist klein, aber es sind einige bedeutende Änderungen erforderlich, damit die App reibungslos läuft.
# Base image https://hub.docker.com/u/rocker/
FROM rocker/shiny:latest
# system libraries of general use
## install debian packages
RUN apt-get update -qq && apt-get -y --no-install-recommends install \
libxml2-dev \
libcairo2-dev \
libsqlite3-dev \
libmariadbd-dev \
libpq-dev \
libssh2-1-dev \
unixodbc-dev \
libcurl4-openssl-dev \
libssl-dev
## update system libraries
RUN apt-get update && \
apt-get upgrade -y && \
apt-get clean
# copy necessary files
## renv.lock file
COPY /example-app/renv.lock ./renv.lock
## app folder
COPY /example-app ./app
# install renv & restore packages
RUN Rscript -e 'install.packages("renv")'
RUN Rscript -e 'renv::restore()'
# expose port
EXPOSE 3838
# run app on container start
CMD ["R", "-e", "shiny::runApp('/app', host = '0.0.0.0', port = 3838)"]Das Base-Image
Die erste Änderung betrifft das Base-Image. Da wir hier eine ShinyApp verdockern, können wir uns eine Menge Arbeit ersparen, indem wir das Base Image „rocker/shiny“ verwenden. Dieses Image kümmert sich um die notwendigen Dependencies für die Ausführung einer ShinyApp und enthält mehrere R-Pakete, die bereits vorinstalliert sind.
Erforderliche Dateien
Es ist natürlich notwendig, alle relevanten Skripte und Dateien für deine ShinyApp in dein Docker-Image einzubauen. Das Dockerfile erledigt genau das, indem es den gesamten Ordner, der die Anwendung enthält, in das Image kopiert.
Um die benötigten R-Pakete in ihrer korrekten Version in das Docker-Image zu installieren, verwenden wir am besten renv. Deshalb kopieren wir zuerst die Datei renv.lock separat in das Image. Das renv-Paket muss ebenfalls separat installiert werden, indem wir die Fähigkeit des Dockerfiles nutzen, mithilfe von RUN Rscript -e R-Code auszuführen. Diese Paketinstallation erlaubt es uns renv direkt aufzurufen und die kopierte Paketbibliothek in renv.lock innerhalb des Images mit renv::restore() wiederherzustellen. Dadurch wird die gesamte Paketbibliothek im Docker-Image installiert, mit genau der gleichen Version und dem gleichen Quellcode aller Pakete wie in der lokalen Projektumgebung. Und all dies mit nur ein paar Zeilen Code in unserem Dockerfile.
Starten der App zur Laufzeit
Ganz am Ende des Dockerfiles weisen wir den Container an, den folgenden R-Befehl auszuführen:
shiny::runApp('/app', host = '0.0.0.0', port = 3838)Das erste Argument legt den Dateipfad zu den benötigten Skripten fest, welche in diesem Fall unter ./app abgelegt sind. Für den exponierten Port habe ich 3838 gewählt. Dies entspricht der Standardeinstellung für RStudio Server, kann aber nach Belieben geändert werden.
Wenn der letzte Befehl ausgeführt wird, startet jeder Container, der auf diesem Image basiert, die betreffende Anwendung automatisch zur Laufzeit (und schließt sie natürlich wieder, wenn sie beendet wurde).
Der letzte Schliff
Mit dem fertigen Dockerfile fehlen nur noch wenige Schritte bis zum laufenden Docker-Container. Dazu muss zunächst noch das Image erstellt werden, um anschließend einen Container basierend auf diesem Image zu starten.
Erstellen des Images
Also öffnen wir das Terminal, navigieren zu dem Ordner, der unser neues Dockerfile enthält und starten den Erstellungsprozess des Images:
docker build -t my-shinyapp-image . Starten eines Containers
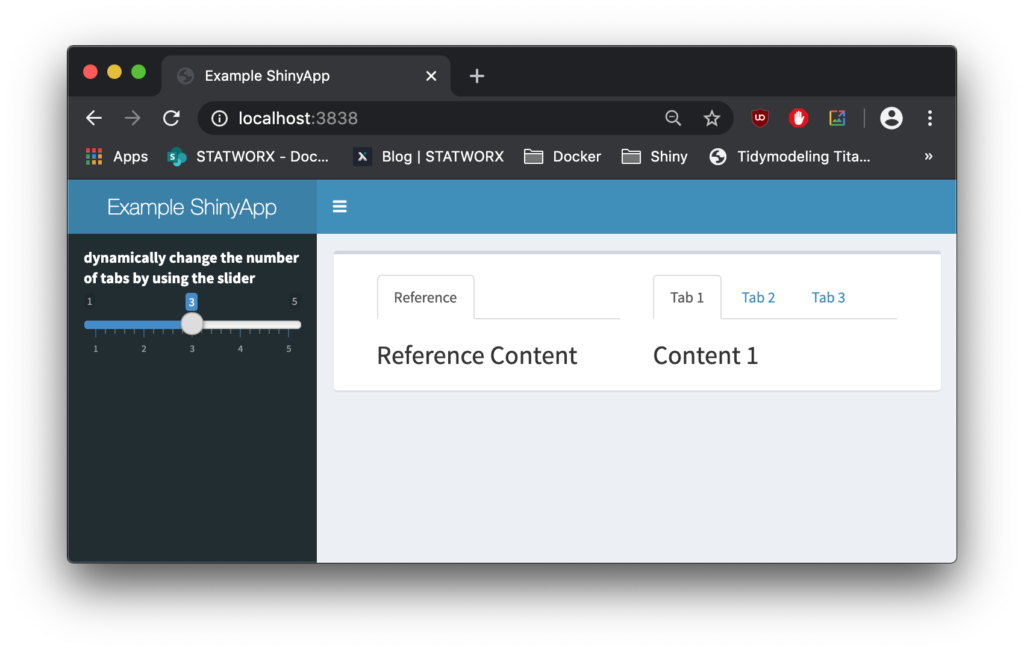
Nachdem dieser Prozess abgeschlossen ist, können wir nun unser neu erstelltes Image testen, indem wir einen Container starten:
docker run -d --rm -p 3838:3838 my-shinyapp-imageUnd da ist sie, die ShinyApp läuft auf localhost:3838!
Ausblick
Jetzt, wo die ShinyApp in einem Docker-Container läuft, ist sie bereit für den Einsatz. Die Containerisierung unserer App macht diesen Prozess bereits sehr viel einfacher. Es gibt jedoch noch weitere Tools, die wir einsetzen, um Sicherheit, Skalierbarkeit und nahtloses Deployment auf dem neuesten Stand der Technik zu gewährleisten. Im nächsten Beitrag erwartet dich deshalb eine Einführung in ShinyProxy, womit du noch tiefer in das Spektrum an Möglichkeiten von RShiny und Docker eintauchen kannst.
Did you know, that you can transform plain old static ggplot graphs to animated ones? Well, you can with the help of the package gganimate by RStudio’s Thomas Lin Pedersen and David Robinson and the results are amazing! My STATWORX colleagues and I are very impressed how effortless all kinds of geoms are transformed to suuuper smooth animations. That’s why in this post I will provide a short overview of some of the wonderful functionalities of gganimate, I hope you’ll enjoy them as much as we do!
Since Valentine’s Day is just around the corner, we’re going to explore the Speed Dating Experiment dataset compiled by Columbia Business School professors Ray Fisman and Sheena Iyengar. Hopefully, we’ll learn about gganimate as well as how to find our Valentine. If you like, you can download the data from Kaggle.
Defining the basic animation: transition_*
How are static plots put into motion? Essentially, gganimate creates data subsets, which are plotted individually and constitute the substantial frames, which, when played consecutively, create the basic animation. The results of gganimate are so seamless because gganimate takes care of the so-called tweening for us by calculating data points for transition frames displayed in-between frames with actual input data.
The transition_* functions define how the data subsets are derived and thus define the general character of any animation. In this blogpost we’re going to explore three types of transitions: transition_states(), transition_reveal() and transition_filter(). But let’s start at the beginning.
We’ll start with transition_states(). Here the data is split into subsets according to the categories of the variable provided to the states argument. If several rows of a dataset pertain to the same unit of observation and should be identifiable as such, a grouping variable defining the observation units needs to be supplied. Alternatively, an identifier can be mapped to any other aesthetic.
Please note, to ensure the readability of this post, all text concerning the interpretation of the speed dating data is written in italics. If you’re not interested in that part you simply can skip those paragraphs. For the data prep, I’d like to refer you to my GitHub.
First, we’re going to explore what the participants of the Speed Dating Experiment look for in a partner. Participants were asked to rate the importance of attributes in a potential date by allocating a budget of 100 points to several characteristics, with higher values denoting a higher importance. The participants were asked to rate the attributes according to their own views. Further, the participants were asked to rate the same attributes according to the presumed wishes of their same-sex peers, meaning they allocated the points in the way they supposed their average same-sex peer would do.
We’re going to plot all of these ratings (x-axis) for all attributes (y-axis). Since we want to compare the individual wishes to the individually presumed wishes of peers, we’re going to transition between both sets of ratings. Color always indicates the personal wishes of a participant. A given bubble indicates the rating of one specific participant for a given attribute, switching between one’s own wishes and the wishes assumed for peers.
## Static Plot
# ...characteristic vs. (presumed) rating...
# ...color&size mapped to own rating, grouped by ID
plot1 <- ggplot(df_what_look_for,
aes(x = value,
y = variable,
color = own_rating, # bubbels are always colord according to own whishes
size = own_rating,
group = iid)) + # identifier of observations across states
geom_jitter(alpha = 0.5, # to reduce overplotting: jitttering & alpha
width = 5) +
scale_color_viridis(option = "plasma", # use virdis' plasma scale
begin = 0.2, # limit range of used hues
name = "Own Rating") +
scale_size(guide = FALSE) + # no legend for size
labs(y = "", # no axis label
x = "Allocation of 100 Points", # x-axis label
title = "Importance of Characteristics for Potential Partner") +
theme_minimal() + # apply minimal theme
theme(panel.grid = element_blank(), # remove all lines of plot raster
text = element_text(size = 16)) # increase font size
## Animated Plot
plot1 +
transition_states(states = rating) # animate contrast subsets acc. to variable rating 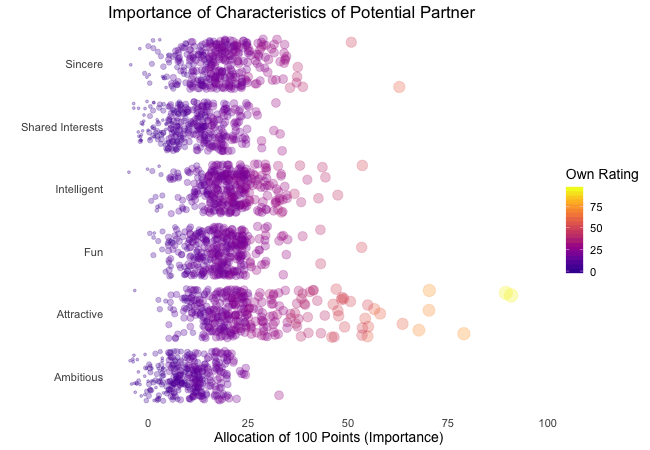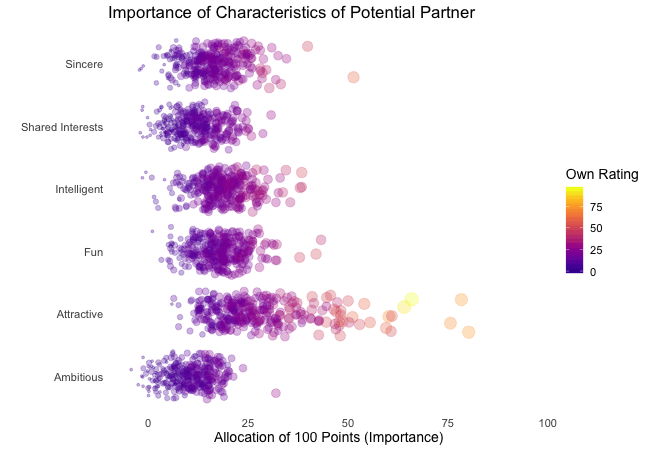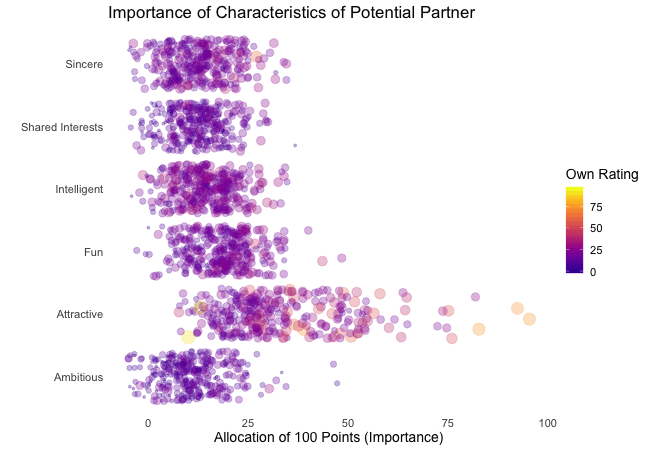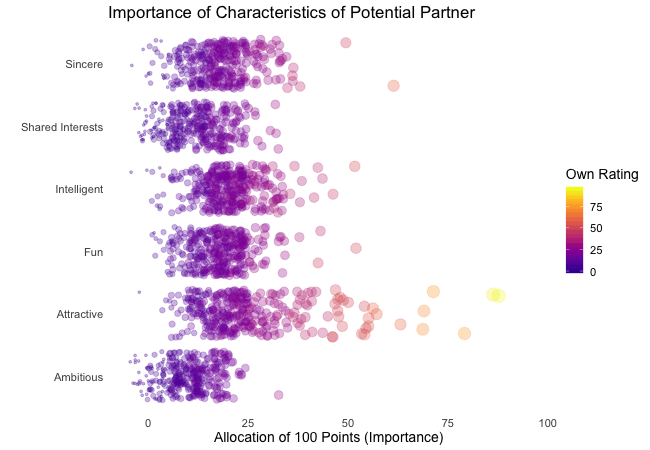
First off, if you’re a little confused which state is which, please be patient, we’ll explore dynamic labels in the section about ‚frame variables‘.
It’s apparent that different people look for different things in a partner. Yet attractiveness is often prioritized over other qualities. But the importance of attractiveness varies most strongly of all attributes between individuals. Interestingly, people are quite aware that their peer’s ratings might differ from their own views. Further, especially the collective presumptions (= the mean values) about others are not completely off, but of higher variance than the actual ratings.
So there is hope for all of us that somewhere out there somebody is looking for someone just as ambitious or just as intelligent as ourselves. However, it’s not always the inner values that count.
gganimate allows us to tailor the details of the animation according to our wishes. With the argument transition_length we can define the relative length of the transition from one to the other real subsets of data takes and with state_length how long, relatively speaking, each subset of original data is displayed. Only if the wrap argument is set to TRUE, the last frame will get morphed back into the first frame of the animation, creating an endless and seamless loop. Of course, the arguments of different transition functions may vary.
## Animated Plot
# ...replace default arguments
plot1 +
transition_states(states = rating,
transition_length = 3, # 3/4 of total time for transitions
state_length = 1, # 1/4 of time to display actual data
wrap = FALSE) # no endless loop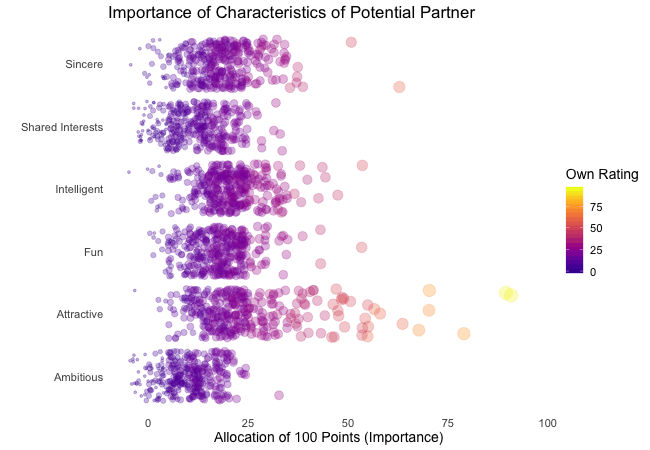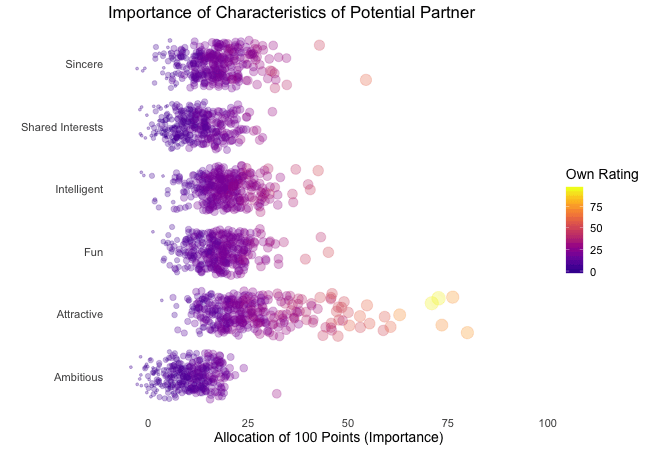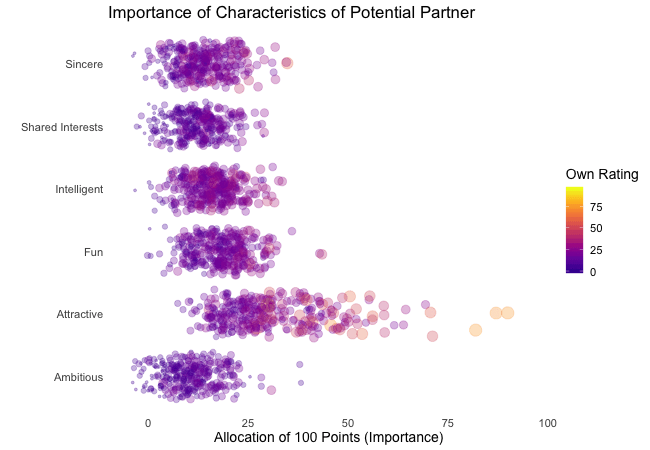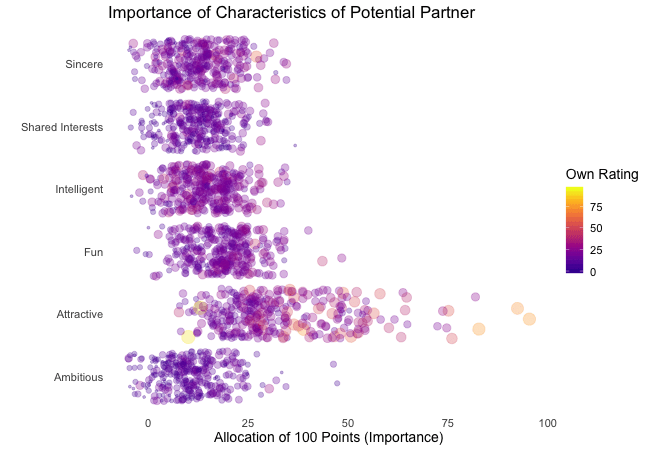
Styling transitions: ease_aes
As mentioned before, gganimate takes care of tweening and calculates additional data points to create smooth transitions between successively displayed points of actual input data. With ease_aes we can control which so-called easing function is used to ‚morph‘ original data points into each other. The default argument is used to declare the easing function for all aesthetics in a plot. Alternatively, easing functions can be assigned to individual aesthetics by name. Amongst others quadric, cubic , sine and exponential easing functions are available, with the linear easing function being the default. These functions can be customized further by adding a modifier-suffix: with -in the function is applied as-is, with -out the function is reversely applied with -in-out the function is applied as-is in the first half of the transition and reversed in the second half.
Here I played around with an easing function that models the bouncing of a ball.
## Animated Plot
# ...add special easing function
plot1 +
transition_states(states = rating) +
ease_aes("bounce-in") # bouncy easing function, as-is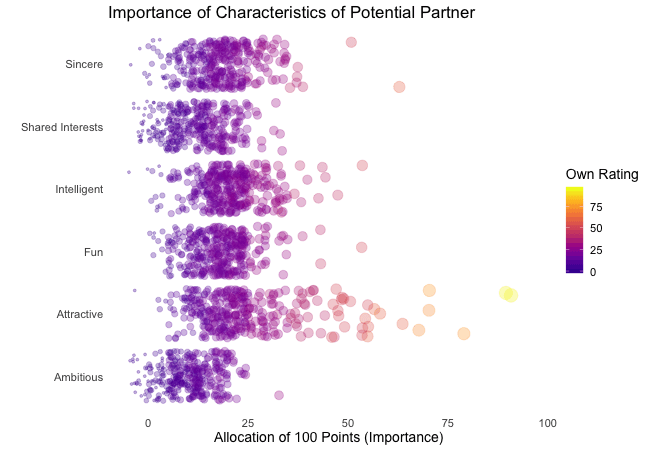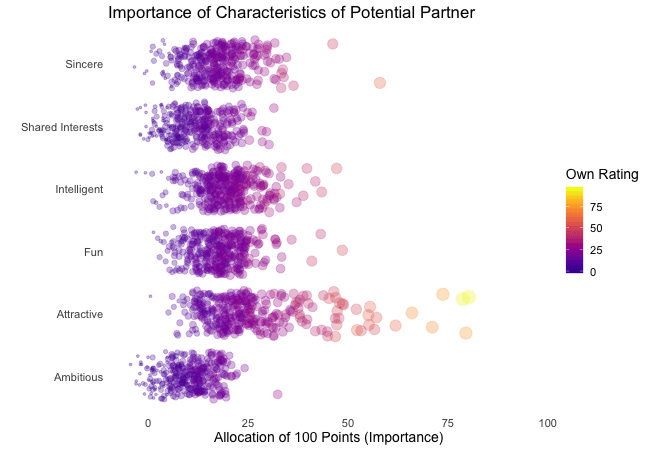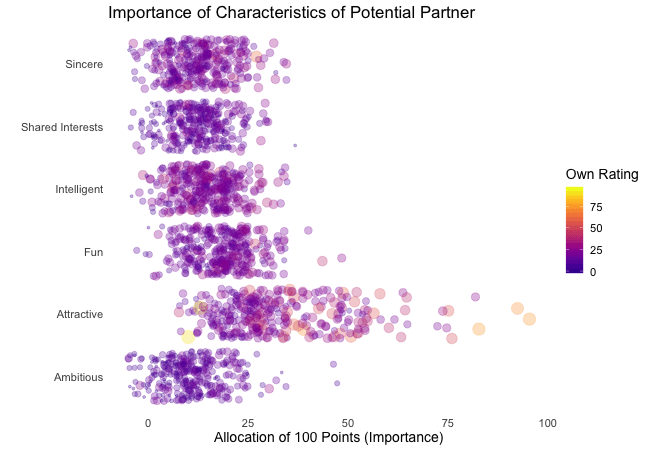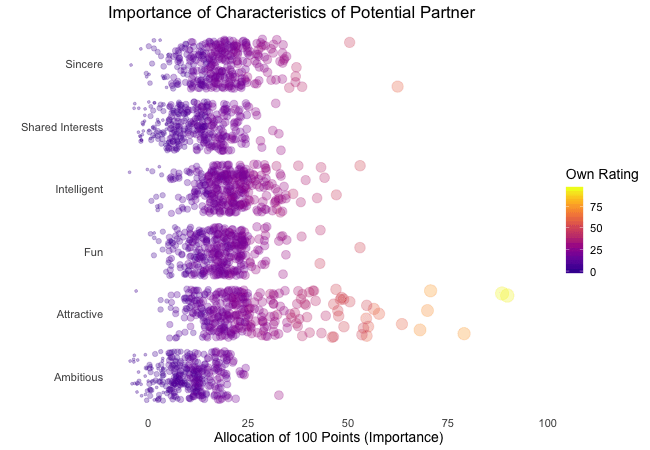
Dynamic labelling: {frame variables}
To ensure that we, mesmerized by our animations, do not lose the overview gganimate provides so-called frame variables that provide metadata about the animation as a whole or the previous/current/next frame. The frame variables – when wrapped in curly brackets – are available for string literal interpretation within all plot labels. For example, we can label each frame with the value of the states variable that defines the currently (or soon to be) displayed subset of actual data:
## Animated Plot
# ...add dynamic label: subtitle with current/next value of states variable
plot1 +
labs(subtitle = "{closest_state}") + # add frame variable as subtitle
transition_states(states = rating) 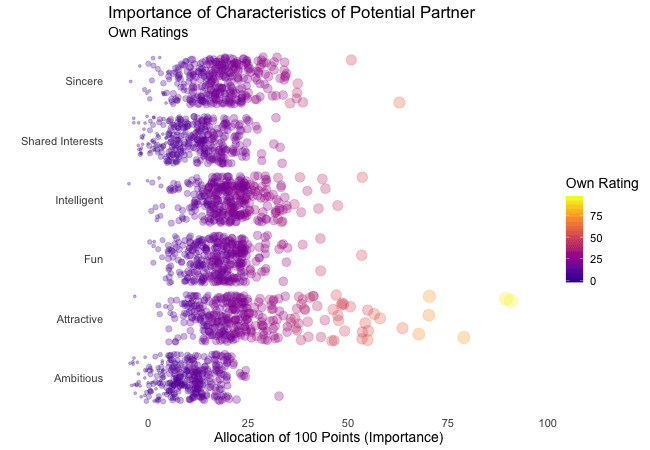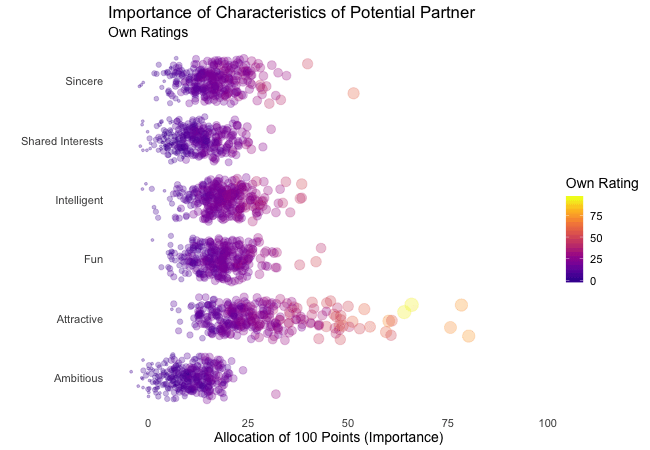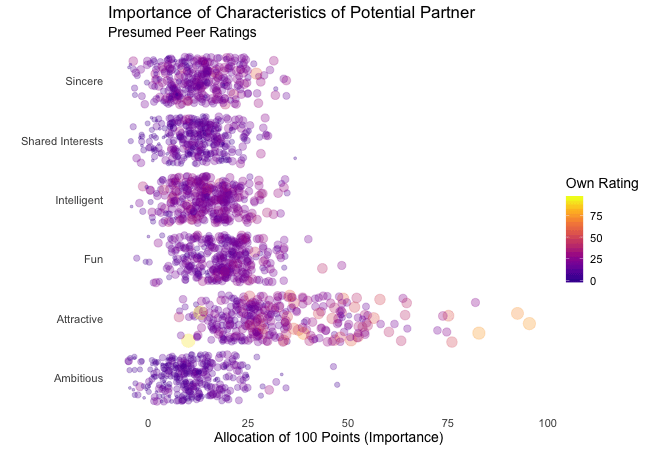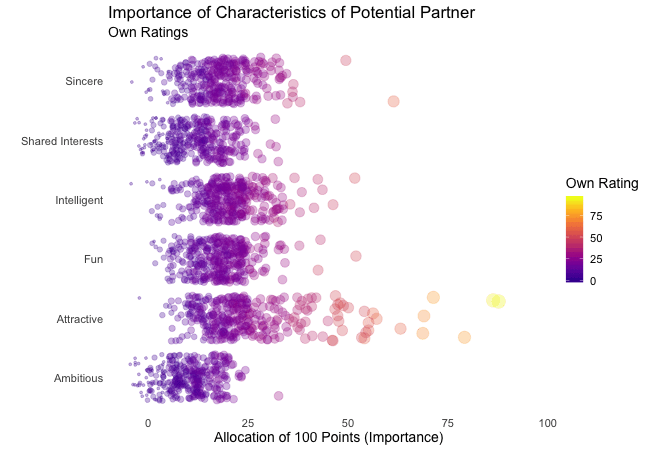
The set of available variables depends on the transition function. To get a list of frame variables available for any animation (per default the last one) the frame_vars() function can be called, to get both the names and values of the available variables.
Indicating previous data: shadow_*
To accentuate the interconnection of different frames, we can apply one of gganimates ’shadows‘. Per default shadow_null() i.e. no shadow is added to animations. In general, shadows display data points of past frames in different ways: shadow_trail() creates a trail of evenly spaced data points, while shadow_mark() displays all raw data points.
We’ll use shadow_wake() to create a little ‚wake‘ of past data points which are gradually shrinking and fading away. The argument wake_length allows us to set the length of the wake, relative to the total number of frames. Since the wakes overlap, the transparency of geoms might need adjustment. Obviously, for plots with lots of data points shadows can impede the intelligibility.
plot1B + # same as plot1, but with alpha = 0.1 in geom_jitter
labs(subtitle = "{closest_state}") +
transition_states(states = rating) +
shadow_wake(wake_length = 0.5) # adding shadow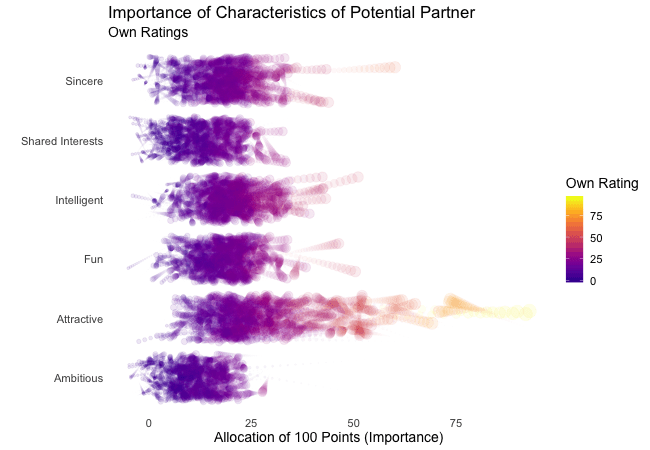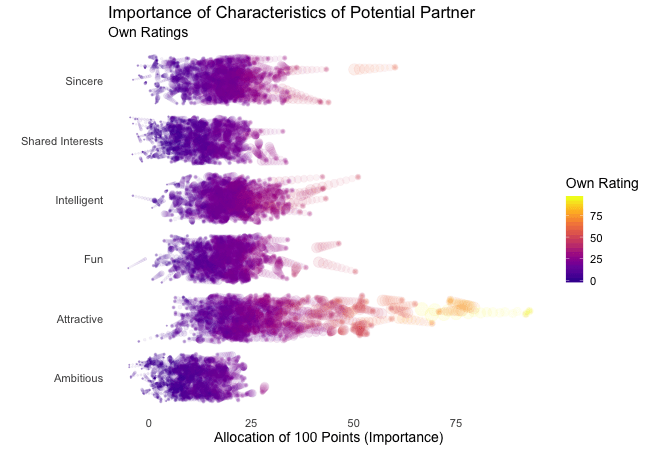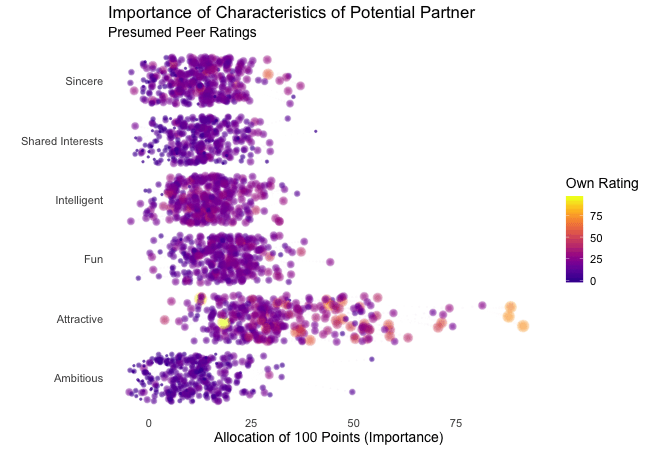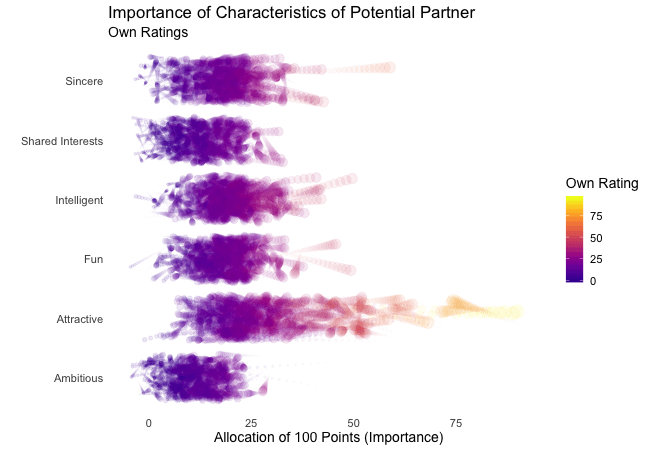
The benefits of transition_*
While I simply love the visuals of animated plots, I think they’re also offering actual improvement. I feel transition_states compared to facetting has the advantage of making it easier to track individual observations through transitions. Further, no matter how many subplots we want to explore, we do not need lots of space and clutter our document with thousands of plots nor do we have to put up with tiny plots.
Similarly, e.g. transition_reveal holds additional value for time series by not only mapping a time variable on one of the axes but also to actual time: the transition length between the individual frames displays of actual input data corresponds to the actual relative time differences of the mapped events. To illustrate this, let’s take a quick look at the ’success‘ of all the speed dates across the different speed dating events:
## Static Plot
# ... date of event vs. interest in second date for women, men or couples
plot2 <- ggplot(data = df_match,
aes(x = date, # date of speed dating event
y = count, # interest in 2nd date
color = info, # which group: women/men/reciprocal
group = info)) +
geom_point(aes(group = seq_along(date)), # needed, otherwise transition dosen't work
size = 4, # size of points
alpha = 0.7) + # slightly transparent
geom_line(aes(lty = info), # line type according to group
alpha = 0.6) + # slightly transparent
labs(y = "Interest After Speed Date",
x = "Date of Event",
title = "Overall Interest in Second Date") +
scale_linetype_manual(values = c("Men" = "solid", # assign line types to groups
"Women" = "solid",
"Reciprocal" = "dashed"),
guide = FALSE) + # no legend for linetypes
scale_y_continuous(labels = scales::percent_format(accuracy = 1)) + # y-axis in %
scale_color_manual(values = c("Men" = "#2A00B6", # assign colors to groups
"Women" = "#9B0E84",
"Reciprocal" = "#E94657"),
name = "") +
theme_minimal() + # apply minimal theme
theme(panel.grid = element_blank(), # remove all lines of plot raster
text = element_text(size = 16)) # increase font size
## Animated Plot
plot2 +
transition_reveal(along = date) 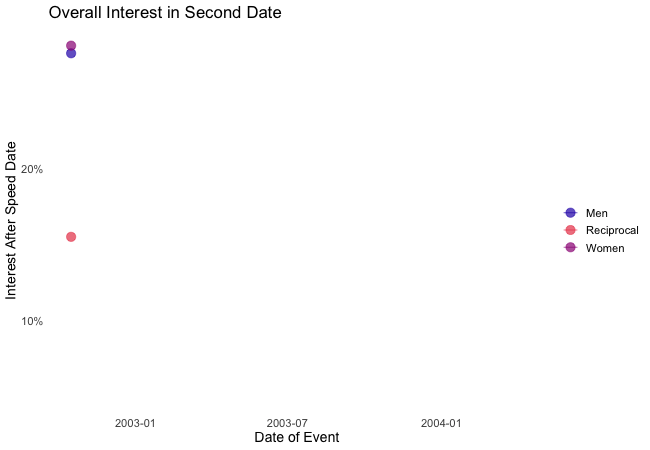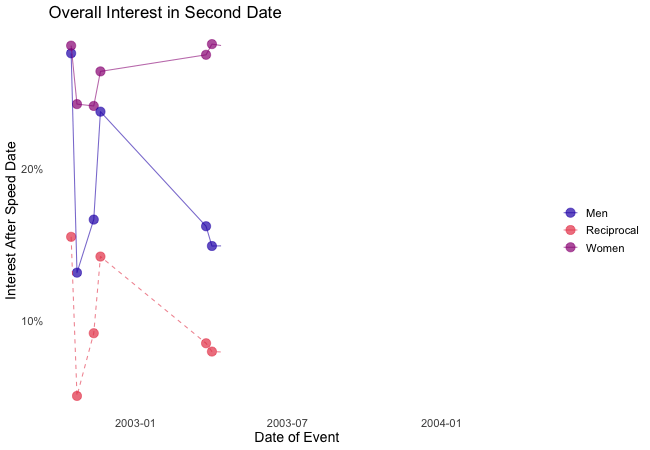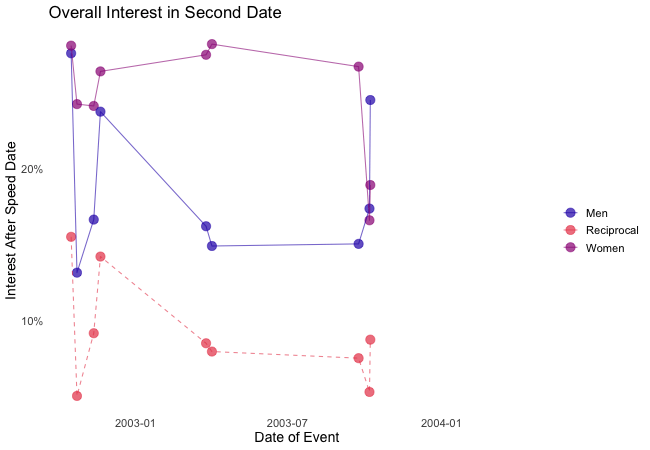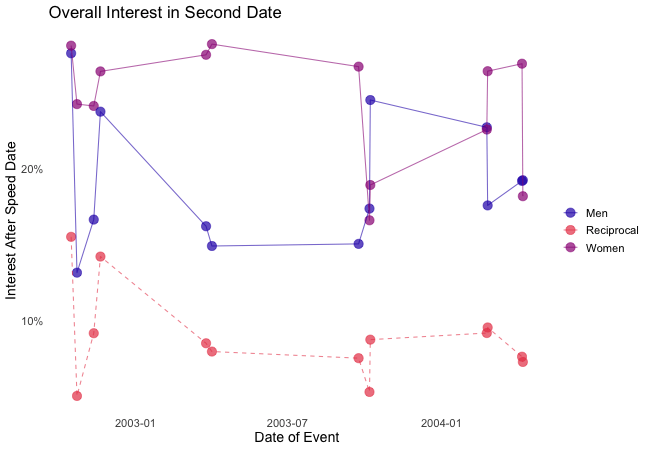
Displayed are the percentages of women and men who were interested in a second date after each of their speed dates as well as the percentage of couples in which both partners wanted to see each other again.
Most of the time, women were more interested in second dates than men. Further, the attraction between dating partners often didn’t go both ways: the instances in which both partners of a couple wanted a second date always were far more infrequent than the general interest of either men and women. While it’s hard to identify the most romantic time of the year, according to the data there seemed to be a slack in romance in early autumn. Maybe everybody still was heartbroken over their summer fling? Fortunately, Valentine’s Day is in February.
Another very handy option is transition_filter(), it’s a great way to present selected key insights of your data exploration. Here the animation browses through data subsets defined by a series of filter conditions. It’s up to you which data subsets you want to stage. The data is filtered according to logical statements defined in transition_filter(). All rows for which a statement holds true are included in the respective subset. We can assign names to the logical expressions, which can be accessed as frame variables. If the keep argument is set to TRUE, the data of previous frames is permanently displayed in later frames.
I want to explore, whether one’s own characteristics relate to the attributes one looks for in a partner. Do opposites attract? Or do birds of a feather (want to) flock together?
Displayed below are the importances the speed dating participants assigned to different attributes of a potential partner. Contrasted are subsets of participants, who were rated especially funny, attractive, sincere, intelligent or ambitious by their speed dating partners. The rating scale went from 1 = low to 10 = high, thus I assume value of >7 to be rather outstanding.
## Static Plot (without geom)
# ...importance ratings for different attributes
plot3 <- ggplot(data = df_ratings,
aes(x = variable, # different attributes
y = own_rating, # importance regarding potential partner
size = own_rating,
color = variable, # different attributes
fill = variable)) +
geom_jitter(alpha = 0.3) +
labs(x = "Attributes of Potential Partner", # x-axis label
y = "Allocation of 100 Points (Importance)", # y-axis label
title = "Importance of Characteristics of Potential Partner", # title
subtitle = "Subset of {closest_filter} Participants") + # dynamic subtitle
scale_color_viridis_d(option = "plasma", # use viridis scale for color
begin = 0.05, # limit range of used hues
end = 0.97,
guide = FALSE) + # don't show legend
scale_fill_viridis_d(option = "plasma", # use viridis scale for filling
begin = 0.05, # limit range of used hues
end = 0.97,
guide = FALSE) + # don't show legend
scale_size_continuous(guide = FALSE) + # don't show legend
theme_minimal() + # apply minimal theme
theme(panel.grid = element_blank(), # remove all lines of plot raster
text = element_text(size = 16)) # increase font size
## Animated Plot
# ...show ratings for different subsets of participants
plot3 +
geom_jitter(alpha = 0.3) +
transition_filter("More Attractive" = Attractive > 7, # adding named filter expressions
"Less Attractive" = Attractive <= 7,
"More Intelligent" = Intelligent > 7,
"Less Intelligent" = Intelligent <= 7,
"More Fun" = Fun > 7,
"Less Fun" = Fun <= 5) 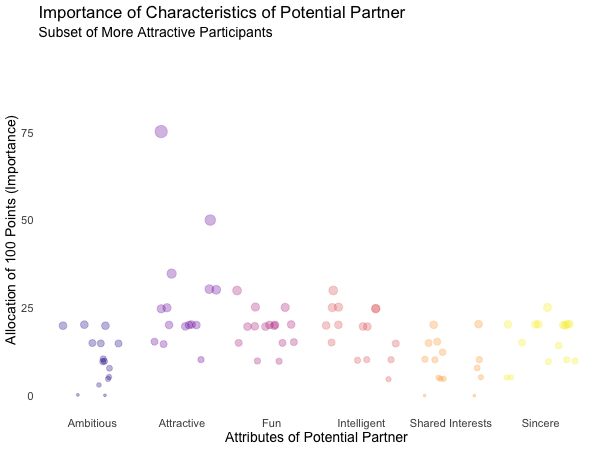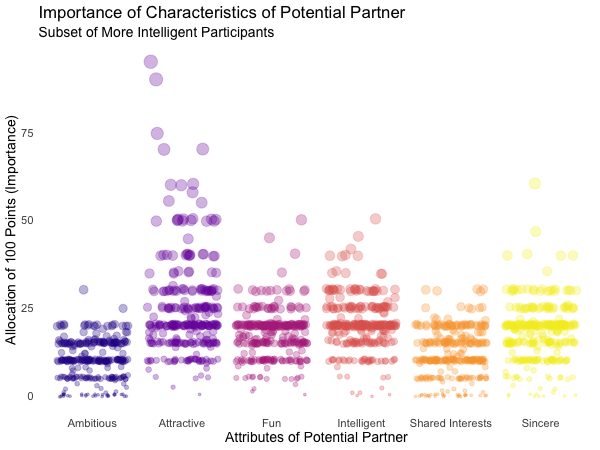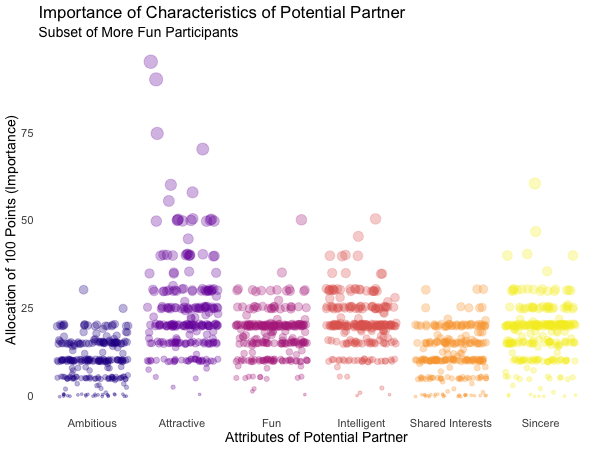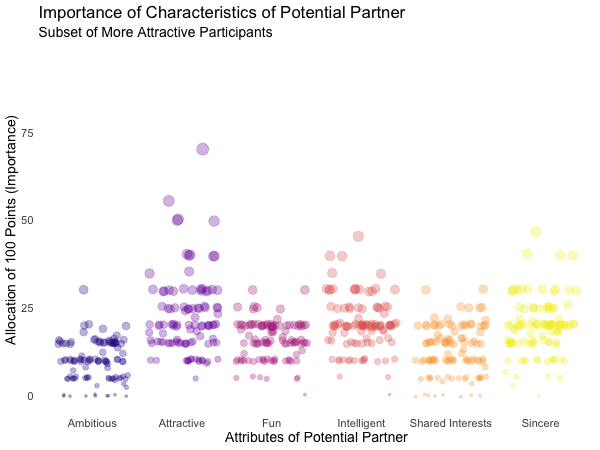
Of course, the number of extraordinarily attractive, intelligent or funny participants is relatively low. Surprisingly, there seem to be little differences between what the average low vs. high scoring participants look for in a partner. Rather the lower scoring group includes more people with outlying expectations regarding certain characteristics. Individual tastes seem to vary more or less independently from individual characteristics.
Styling the (dis)appearance of data: enter_* / exit_*
Especially if displayed subsets of data do not or only partially overlap, it can be favorable to underscore this visually. A good way to do this are the enter_*() and exit_*() functions, which enable us to style the entry and exit of data points, which do not persist between frames.
There are many combinable options: data points can simply (dis)appear (the default), fade (enter_fade()/exit_fade()), grow or shrink (enter_grow()/exit_shrink()), gradually change their color (enter_recolor()/exit_recolor()), fly (enter_fly()/exit_fly()) or drift (enter_drift()/exit_drift()) in and out.
We can use these stylistic devices to emphasize changes in the databases of different frames. I used exit_fade() to let further not included data points gradually fade away while flying them out of the plot area on a vertical route (y_loc = 100), data points re-entering the sample fly in vertically from the bottom of the plot (y_loc = 0):
## Animated Plot
# ...show ratings for different subsets of participants
plot3 +
geom_jitter(alpha = 0.3) +
transition_filter("More Attractive" = Attractive > 7, # adding named filter expressions
"Less Attractive" = Attractive <= 7,
"More Intelligent" = Intelligent > 7,
"Less Intelligent" = Intelligent <= 7,
"More Fun" = Fun > 7,
"Less Fun" = Fun <= 5) +
enter_fly(y_loc = 0) + # entering data: fly in vertically from bottom
exit_fly(y_loc = 100) + # exiting data: fly out vertically to top...
exit_fade() # ...while color is fading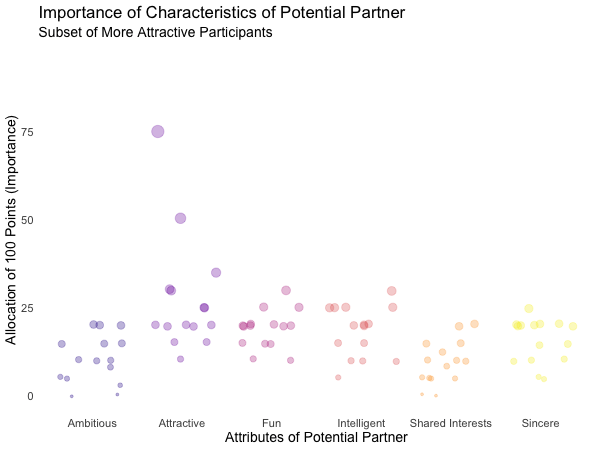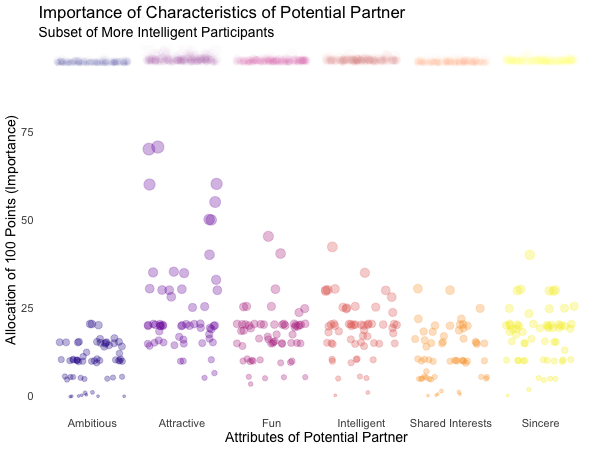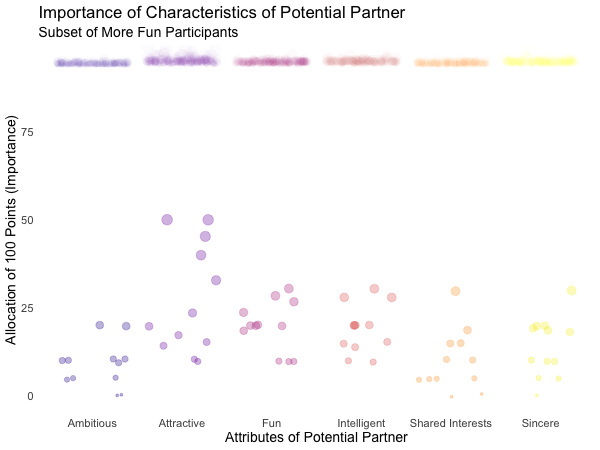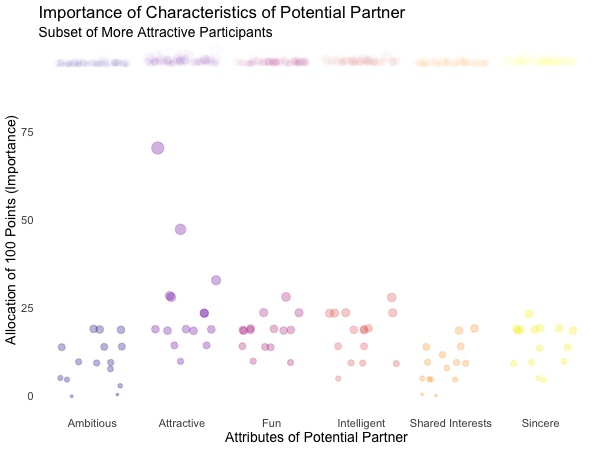
Finetuning and saving: animate() & anim_save()
Gladly, gganimate makes it very easy to finalize and save our animations. We can pass our finished gganimate object to animate() to, amongst other things, define the number of frames to be rendered (nframes) and/or the rate of frames per second (fps) and/or the number of seconds the animation should last (duration). We also have the option to define the device in which the individual frames are rendered (the default is device = “png”, but all popular devices are available). Further, we can define arguments that are passed on to the device, like e.g. width or height. Note, that simply printing an gganimateobject is equivalent to passing it to animate() with default arguments. If we plan to save our animation the argument renderer, is of importance: the function anim_save() lets us effortlessly save any gganimate object, but only so if it was rendered using one of the functions magick_renderer() or the default gifski_renderer().
The function anim_save()works quite straightforward. We can define filename and path (defaults to the current working directory) as well as the animation object (defaults to the most recently created animation).
# create a gganimate object
gg_animation <- plot3 +
transition_filter("More Attractive" = Attractive > 7,
"Less Attractive" = Attractive <= 7)
# adjust the animation settings
animate(gg_animation,
width = 900, # 900px wide
height = 600, # 600px high
nframes = 200, # 200 frames
fps = 10) # 10 frames per second
# save the last created animation to the current directory
anim_save("my_animated_plot.gif")Conclusion (and a Happy Valentine’s Day)
I hope this blog post gave you an idea, how to use gganimate to upgrade your own ggplots to beautiful and informative animations. I only scratched the surface of gganimates functionalities, so please do not mistake this post as an exhaustive description of the presented functions or the package. There is much out there for you to explore, so don’t wait any longer and get started with gganimate!
But even more important: don’t wait on love. The speed dating data shows that most likely there’s someone out there looking for someone just like you. So from everyone here at STATWORX: Happy Valentine’s Day!

## 8 bit heart animation
animation2 <- plot(data = df_eight_bit_heart %>% # includes color and x/y position of pixels
dplyr::mutate(id = row_number()), # create row number as ID
aes(x = x,
y = y,
color = color,
group = id)) +
geom_point(size = 18, # depends on height & width of animation
shape = 15) + # square
scale_color_manual(values = c("black" = "black", # map values of color to actual colors
"red" = "firebrick2",
"dark red" = "firebrick",
"white" = "white"),
guide = FALSE) + # do not include legend
theme_void() + # remove everything but geom from plot
transition_states(-y, # reveal from high to low y values
state_length = 0) +
shadow_mark() + # keep all past data points
enter_grow() + # new data grows
enter_fade() # new data starts without color
animate(animation2,
width = 250, # depends on size defined in geom_point
height = 250, # depends on size defined in geom_point
end_pause = 15) # pause at end of animation
Die Kreuzvalidierung ist eine weit verbreitete Technik zur Bewertung der Generalisierbarkeit eines Machine-Learning-Modells. Hier bei STATWORX diskutieren wir oft über Evaluationsmetriken und darüber, wie wir sie effizient in unseren Data-Science-Workflow einbauen können. In diesem Blog-Beitrag stelle ich die Grundlagen der Kreuzvalidierung vor, gebe Hinweise zur Optimierung ihrer Parameter und zeige, wie man sie auf effiziente Weise von Grund auf selbst erstellen kann.
Grundlagen der Modellbewertung und Kreuzvalidierung
Die Kreuzvalidierung ist eine Technik zur Evaluation von Machine Learning-Modellen. Die zentrale Intuition hinter dieser Modellevaluierung besteht darin, herauszufinden, ob das trainierte Modell verallgemeinerbar ist, d. h., ob die Vorhersagekraft, die wir beim Training beobachten, auch bei ungesehenen Daten zu erwarten ist. Wir könnten das Modell direkt mit den Daten füttern, für die es entwickelt wurde, d. h. die es vorhersagen soll. Aber auch dann gibt es für uns keine Möglichkeit, zu wissen oder zu überprüfen, ob die Vorhersagen zutreffend sind.
Natürlich möchten wir eine Art Benchmark der Generalisierbarkeit unseres Modells haben, bevor wir es in die Produktion überführen. Daher besteht die Idee darin, die vorhandenen Trainingsdaten in einen tatsächlichen Trainingsdatensatz und einen Holdout-Testdatensatz aufzuteilen, die nicht zum Training verwendet wird und als „ungesehene“ Daten dient. Da dieser Testdatensatz Teil der ursprünglichen Trainingsdaten ist, haben wir eine ganze Reihe von „richtigen“ Ergebnissen, die wir überprüfen können. Zur Bewertung der Modellleistung kann dann eine geeignete Fehlermetrik wie der Root Mean Squared Error (RMSE) oder der Mean Absolute Percentage Error (MAPE) verwendet werden. Bei der Wahl der geeigneten Bewertungsmetrik ist jedoch Vorsicht geboten, da es Tücken gibt (wie in diesem Blogbeitrag meines Kollegen Jan beschrieben).
Bei vielen Algorithmen für Machine Learning können Hyperparameter angegeben werden, z. B. die Anzahl der Bäume in einem Random Forest. Die Kreuzvalidierung kann auch zum Tuning der Hyperparameter eines Modells genutzt werden, indem der Generalisierungsfehler verschiedener Modellspezifikationen verglichen wird.
Allgemeine Ansätze zur Modellevaluation
Es gibt Dutzende Techniken zur Modellevaluierung, die immer einen Kompromiss zwischen Varianz, Verzerrung und Rechenzeit darstellen. Bei der Bewertung eines Modells ist es wichtig, diese Kompromisse zu kennen, da die Wahl der geeigneten Technik in hohem Maße von der Problemstellung und den beobachteten Daten abhängt. Ich werde dieses Thema behandeln, nachdem ich zwei der gebräuchlichsten Techniken zur Modellevaluierung vorgestellt habe: den Train-Test-Split und die k-fache Kreuzvalidierung.
Bei der ersten Methode werden die Trainingsdaten nach dem Zufallsprinzip in eine Trainings- und eine Testeinheit aufgeteilt (Abbildung 1), wobei in der Regel ein großer Teil der Daten als Trainingsset beibehalten wird. In der Literatur werden am häufigsten Verhältnisse von 70/30 oder 80/20 verwendet, wobei das genaue Verhältnis von der Größe der Daten abhängt.
Der Nachteil dieses Ansatzes ist, dass diese einmalige zufällige Aufteilung dazu führen kann, dass die Daten in zwei sehr unausgewogene Teile aufgeteilt werden, was zu verzerrten Schätzungen des Generalisierungsfehlers führt. Dies ist besonders kritisch, wenn man nur über begrenzte Daten verfügt, da einige Merkmale oder Muster vollständig im Testteil landen könnten. In einem solchen Fall hat das Modell keine Chance, sie zu lernen, und seine Leistung wird möglicherweise unterschätzt.

Eine robustere Alternative ist die sogenannte k-fache Kreuzvalidierung (Abbildung 2). Hier werden die Daten gemischt und dann nach dem Zufallsprinzip in ![]() Teilmengen aufgeteilt. Der Hauptvorteil gegenüber dem Train-Test-Split besteht darin, dass jede der
Teilmengen aufgeteilt. Der Hauptvorteil gegenüber dem Train-Test-Split besteht darin, dass jede der ![]() Partitionen iterativ als Testdatensatz (d. h. Validierungsmenge) verwendet wird, wobei die verbleibenden
Partitionen iterativ als Testdatensatz (d. h. Validierungsmenge) verwendet wird, wobei die verbleibenden ![]() -Teile in dieser Iteration als Trainingsdaten dienen. Dieser Vorgang wird
-Teile in dieser Iteration als Trainingsdaten dienen. Dieser Vorgang wird ![]() Mal wiederholt, sodass jede Beobachtung sowohl in den Trainings- als auch in den Testdaten enthalten ist. Die entsprechende Fehlermetrik wird dann einfach als Mittelwert aus allen
Mal wiederholt, sodass jede Beobachtung sowohl in den Trainings- als auch in den Testdaten enthalten ist. Die entsprechende Fehlermetrik wird dann einfach als Mittelwert aus allen ![]() Iterationen berechnet und ergibt den Kreuzvalidierungsfehler.
Iterationen berechnet und ergibt den Kreuzvalidierungsfehler.
Dabei handelt es sich eher um eine Erweiterung des Train-Test-Splits als um eine völlig neue Methode: Das heißt, das Train-Test-Verfahren wird ![]() Mal wiederholt. Wenn
Mal wiederholt. Wenn ![]() z.B. so niedrig wie
z.B. so niedrig wie ![]() gewählt wird, erhält man zwar nur zwei Sets. Aber auch dieser Ansatz ist dem Train-Test-Split immer noch überlegen, da beide Teile iterativ für das Training ausgewählt werden, sodass das Modell die Chance hat, aus allen Daten zu lernen und nicht nur eine zufällige Teilmenge davon. Daher führt dieser Ansatz in der Regel zu robusteren Leistungsschätzungen.
gewählt wird, erhält man zwar nur zwei Sets. Aber auch dieser Ansatz ist dem Train-Test-Split immer noch überlegen, da beide Teile iterativ für das Training ausgewählt werden, sodass das Modell die Chance hat, aus allen Daten zu lernen und nicht nur eine zufällige Teilmenge davon. Daher führt dieser Ansatz in der Regel zu robusteren Leistungsschätzungen.

Wenn man die beiden Abbildungen oben vergleichtt, sieht man, dass eine Aufteilung von Training und Test mit einem Verhältnis von 80/20 einer Iteration einer 5-fachen (d.h. ![]() ) Kreuzvalidierung entspricht, bei der 4/5 der Daten für das Training und 1/5 für die Validierung zurückbehalten werden. Der entscheidende Unterschied ist, dass bei der k-fachen Kreuzvalidierung die Validierungsmenge in jeder der
) Kreuzvalidierung entspricht, bei der 4/5 der Daten für das Training und 1/5 für die Validierung zurückbehalten werden. Der entscheidende Unterschied ist, dass bei der k-fachen Kreuzvalidierung die Validierungsmenge in jeder der ![]() Iterationen verschoben wird. Beachte, dass eine k-fache Kreuzvalidierung robuster ist als die einfache Wiederholung der Aufteilung von Training und Test
Iterationen verschoben wird. Beachte, dass eine k-fache Kreuzvalidierung robuster ist als die einfache Wiederholung der Aufteilung von Training und Test ![]() Mal: Bei der k-fachen Kreuzvalidierung wird die Aufteilung einmal vorgenommen und dann durch die Partitionen iteriert, wohingegen bei der wiederholten Aufteilung von Training und Test die Daten
Mal: Bei der k-fachen Kreuzvalidierung wird die Aufteilung einmal vorgenommen und dann durch die Partitionen iteriert, wohingegen bei der wiederholten Aufteilung von Training und Test die Daten ![]() -mal neu aufgeteilt werden, wodurch möglicherweise einige Daten beim Training ausgelassen werden.
-mal neu aufgeteilt werden, wodurch möglicherweise einige Daten beim Training ausgelassen werden.
Wiederholte CV und LOOCV
Es gibt viele Varianten der k-fachen Kreuzvalidierung. Man kann zum Beispiel auch eine „wiederholte Kreuzvalidierung“ durchführen. Die Idee dahinter ist, dass sobald die Daten einmal in ![]() Partitionen aufgeteilt wurden, diese Aufteilung für das gesamte Verfahren gilt. Auf diese Weise besteht nicht die Gefahr, dass wir zufällig einige Teile ausschließen. Bei der Repeated CV wiederholst du diesen Prozess des Mischens und der zufälligen Aufteilung der Daten in
Partitionen aufgeteilt wurden, diese Aufteilung für das gesamte Verfahren gilt. Auf diese Weise besteht nicht die Gefahr, dass wir zufällig einige Teile ausschließen. Bei der Repeated CV wiederholst du diesen Prozess des Mischens und der zufälligen Aufteilung der Daten in ![]() Teilmengen eine gewisse Anzahl von Malen. Anschließend kannst du den Durchschnitt über die resultierenden Kreuzvalidierungsfehler der einzelnen Durchgänge bilden, um eine globale Leistungsschätzung zu erhalten.
Teilmengen eine gewisse Anzahl von Malen. Anschließend kannst du den Durchschnitt über die resultierenden Kreuzvalidierungsfehler der einzelnen Durchgänge bilden, um eine globale Leistungsschätzung zu erhalten.
Ein weiterer Spezialfall der k-fachen Kreuzvalidierung ist die „Leave One Out Cross-Validation“ (LOOCV), bei der du ![]() setzt. Das heißt, du verwendest in jeder Iteration eine einzige Beobachtung aus deinen Daten als Validierungsteil und die restlichen
setzt. Das heißt, du verwendest in jeder Iteration eine einzige Beobachtung aus deinen Daten als Validierungsteil und die restlichen ![]() Beobachtungen als Trainingsmenge. Das klingt zwar nach einer sehr robusten Version der Kreuzvalidierung, aber von dieser Methode wird im Allgemeinen aus zwei Gründen abgeraten:
Beobachtungen als Trainingsmenge. Das klingt zwar nach einer sehr robusten Version der Kreuzvalidierung, aber von dieser Methode wird im Allgemeinen aus zwei Gründen abgeraten:
- Erstens ist sie in der Regel sehr rechenintensiv. Für die meisten Datensätze, die beim angewandten Machine Learning verwendet werden, ist es weder erstrebenswert noch umsetzbar, das Modell
 Mal zu trainieren (obwohl es für sehr kleine Datensätze nützlich sein kann).
Mal zu trainieren (obwohl es für sehr kleine Datensätze nützlich sein kann). - Zweitens: Selbst wenn man die Rechenleistung (und Zeit) hätte, um diesen Prozess durchzuführen, ist ein weiteres Argument, das von Kritiker:innen von LOOCV aus statistischer Sicht vorgebracht wird, dass der resultierende Kreuzvalidierungsfehler eine hohe Varianz aufweisen kann. Das liegt daran, dass das „Validierungsset“ nur aus einer einzelnen Beobachtung besteht, und je nach Verteilung deiner Daten (und möglichen Ausreißern) kann diese erheblich variieren.
Generell sollte man beachten, dass die Leistung von LOOCV sowohl in der wissenschaftlichen Literatur als auch in der breiteren Machine Learning Community ein etwas kontroverses Thema ist. Daher empfehle ich, sich über diese Debatte zu informieren, wenn man in Erwägung zieht, LOOCV zur Schätzung der Generalisierbarkeit eines Modells zu verwenden (siehe z. B. hier) und ähnliche Beiträge auf StackExchange). Wie so oft könnte die Antwort lauten: „Es kommt darauf an“. Bedenke auf jeden Fall den Rechenaufwand von LOOCV, der kaum von der Hand zu weisen ist (außer du hast einen winzigen Datensatz).
Der Wert von 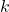 und der Kompromiss zwischen Verzerrung und Varianz
und der Kompromiss zwischen Verzerrung und Varianz
Wenn ![]() nicht (unbedingt) die beste Wahl ist, wie findet man dann einen geeigneten Wert für
nicht (unbedingt) die beste Wahl ist, wie findet man dann einen geeigneten Wert für ![]() ? Es stellt sich heraus, dass die Antwort auf diese Frage auf den berüchtigten Bias-Variance Trade-Off hinausläuft. Warum ist das so?
? Es stellt sich heraus, dass die Antwort auf diese Frage auf den berüchtigten Bias-Variance Trade-Off hinausläuft. Warum ist das so?
Der Wert für ![]() bestimmt, in wie viele Partitionen die Daten unterteilt werden und damit auch die Größe (d.h. die Anzahl der Beobachtungen, die in jedem Teil enthalten sind). Wir wollen
bestimmt, in wie viele Partitionen die Daten unterteilt werden und damit auch die Größe (d.h. die Anzahl der Beobachtungen, die in jedem Teil enthalten sind). Wir wollen ![]() so wählen, dass ein ausreichend großer Teil unserer Daten in der Trainingsmenge verbleibt – schließlich wollen wir nicht zu viele Beobachtungen verschenken, die zum Trainieren unseres Modells verwendet werden könnten. Je höher der Wert von
so wählen, dass ein ausreichend großer Teil unserer Daten in der Trainingsmenge verbleibt – schließlich wollen wir nicht zu viele Beobachtungen verschenken, die zum Trainieren unseres Modells verwendet werden könnten. Je höher der Wert von ![]() ist, desto mehr Beobachtungen werden bei jeder Iteration in die Trainingsmenge aufgenommen.
ist, desto mehr Beobachtungen werden bei jeder Iteration in die Trainingsmenge aufgenommen.
Nehmen wir zum Beispiel an, wir haben 1.200 Beobachtungen in unserem Datensatz, dann würde unser Trainingssatz bei ![]() aus
aus ![]() Beobachtungen bestehen, aber mit
Beobachtungen bestehen, aber mit ![]() würde es 1.050 Beobachtungen umfassen. Je mehr Beobachtungen für das Training verwendet werden, desto eher approximiert man die tatsächliche Leistung des Modells (so als ob es auf dem gesamten Datensatz trainiert worden wäre) und desto geringer ist die Verzerrung – oder Bias – der Fehlerschätzung im Vergleich zu einem kleineren Teil der Daten. Doch mit zunehmendem
würde es 1.050 Beobachtungen umfassen. Je mehr Beobachtungen für das Training verwendet werden, desto eher approximiert man die tatsächliche Leistung des Modells (so als ob es auf dem gesamten Datensatz trainiert worden wäre) und desto geringer ist die Verzerrung – oder Bias – der Fehlerschätzung im Vergleich zu einem kleineren Teil der Daten. Doch mit zunehmendem ![]() nimmt die Größe der Validierungspartition ab, und die Fehlerschätzung reagiert in jeder Iteration empfindlicher auf diese wenigen Datenpunkte, wodurch die Gesamtvarianz steigen kann. Im Grunde genommen geht es darum, zwischen den „Extremen“ des Train-Test-Splits einerseits und LOOCV andererseits zu wählen. Die folgende Abbildung veranschaulicht schematisch (!) die Bias-Variance Performance und den Rechenaufwand der verschiedenen Kreuzvalidierungsmethoden.
nimmt die Größe der Validierungspartition ab, und die Fehlerschätzung reagiert in jeder Iteration empfindlicher auf diese wenigen Datenpunkte, wodurch die Gesamtvarianz steigen kann. Im Grunde genommen geht es darum, zwischen den „Extremen“ des Train-Test-Splits einerseits und LOOCV andererseits zu wählen. Die folgende Abbildung veranschaulicht schematisch (!) die Bias-Variance Performance und den Rechenaufwand der verschiedenen Kreuzvalidierungsmethoden.
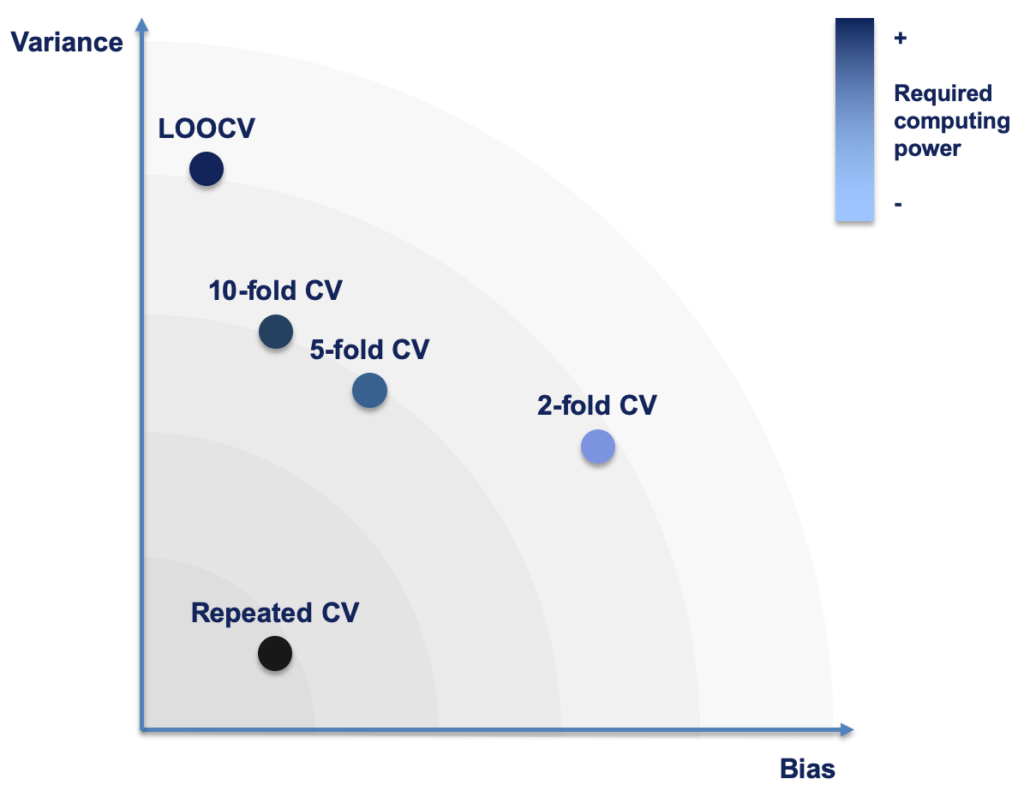
Als Faustregel gilt: Mit höheren Werten für k nimmt die Verzerrung ab und die Varianz zu. Konventionell gelten Werte wie ![]() oder
oder ![]() als guter Kompromiss und sind daher in den meisten Fällen des Machine Learning zum Quasi-Standard geworden.
als guter Kompromiss und sind daher in den meisten Fällen des Machine Learning zum Quasi-Standard geworden.
„These values have been shown empirically to yield test error rate estimates that suffer neither from excessively high bias nor from very high variance.“
James et al. 2013: 184
Wenn du dich nicht besonders für den Prozess der Kreuzvalidierung interessierst, sondern ihn einfach in deinen Data Science Workflow integrieren willst (was ich dir sehr empfehle!), solltest du einen dieser Werte für ![]() wählen und es dabei belassen.
wählen und es dabei belassen.
Implementierung der Kreuzvalidierung in
Apropos Integration der Kreuzvalidierung in deinen täglichen Arbeitsablauf – welche Möglichkeiten gibt es da? Glücklicherweise ist die Kreuzvalidierung ein Standardwerkzeug in beliebten Paketen für Machine Learning, wie z. B. dem Paket caret in R. Hier kannst du die Methode mit der Funktion trainControl festlegen. Im folgenden Skript trainieren wir einen Random Forest mit 10-facher Kreuzvalidierung auf dem iris Datensatz.
library(caret)
set.seed(12345)
inTrain <- createDataPartition(y = iris[["Species"]], p = .7, list = FALSE)
iris.train <- iris[inTrain, ]
iris.test <- iris[- inTrain, ]
fit.control <- caret::trainControl(method = "cv", number = 10)
rf.fit <- caret::train(Species ~ .,
data = iris.train,
method = "rf",
trControl = fit.control)Wir definieren unsere gewünschte Kreuzvalidierungsmethode in der Funktion trainControl, speichern die Ausgabe im Objekt fit.control und übergeben dieses Objekt dann an das Argument trControl der Funktion train. Du kannst die anderen in diesem Beitrag vorgestellten Methoden auf ähnliche Weise definieren:
# Leave-One-Out Cross-validation:
fit.control <- caret::trainControl(method = "LOOCV", number = 10)
# Repeated CV (remember to specify the number of repeats!)
fit.control <- caret::trainControl(method = "repeatedcv", number = 10, repeats = 5)Die altmodische Art: Die 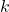 -fold Kreuzvalidierung von Hand implementieren
-fold Kreuzvalidierung von Hand implementieren
Data Science-Projekte können jedoch schnell so komplex werden, dass die vorgefertigten Funktionen in Machine Learning-Paketen nicht mehr geeignet sind. In solchen Fällen musst du den Algorithmus – einschließlich der Kreuzvalidierungstechniken – von Hand implementieren, zugeschnitten auf die spezifischen Projektanforderungen. Ich möchte dir ein provisorisches Skript zeigen, mit dem du eine einfache k-fache Kreuzvalidierung in R von Hand implementieren kannst (wir gehen das Skript hier Schritt für Schritt an; den gesamten Code findest du auf GitHub).
Daten simulieren, Fehlermetrik definieren und 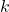 festlegen
festlegen
# devtools::install_github("andrebleier/Xy")
library(tidyverse)
library(Xy)
sim <- Xy(n = 1000,
numvars = c(2,2),
catvars = 0,
cor = c(-0.5, 0.9),
noisevars = 0)
sim_data <- sim[["data"]]
RMSE <- function(f, o){
sqrt(mean((f - o)^2))
}
k <- 5Wir beginnen damit, die benötigten Pakete zu laden und einige Simulationsdaten mit 1.000 Beobachtungen mit dem Paket Xy() zu simulieren, das mein Kollege André entwickelt hat (siehe seinen Blogbeitrag über Regressionsdaten mit Xy simulieren). Da wir eine Art Fehlermaß zur Bewertung der Modellleistung benötigen, definieren wir unsere RMSE-Funktion, die ziemlich einfach ist: Der RMSE ist der Root Mean Squared Error, also die Wurzel aus dem Mittelwert des quadrierten Fehlers, wobei der Fehler die Differenz zwischen den gefitteten (fitted; f) und den beobachteten (observed; o) Werten ist – man kann die Funktion so ziemlich von links nach rechts lesen. Zuletzt geben wir unser k an, das im Beispiel auf den Wert 5 gesetzt ist und als einfacher Integer gespeichert wird.
Aufteilung der Daten
set.seed(12345)
sim_data <- mutate(sim_data,
my.folds = sample(1:k,
size = nrow(sim_data),
replace = TRUE)) Als nächstes unterteilen wir unsere Daten in ![]() folds. Hierfür fügen wir den Daten eine neue Spalte hinzu,
folds. Hierfür fügen wir den Daten eine neue Spalte hinzu,my.folds: Wir ziehen eine Stichprobe (mit Zurücklegen) von 1 bis zum Wert von ![]() , in unserem Fall also 1 bis 5, und fügen jeder Zeile (Beobachtung) in den Daten zufällig eine dieser fünf Zahlen hinzu. Bei 1.000 Beobachtungen sollte jede Zahl etwa 200 Mal zugewiesen werden.
, in unserem Fall also 1 bis 5, und fügen jeder Zeile (Beobachtung) in den Daten zufällig eine dieser fünf Zahlen hinzu. Bei 1.000 Beobachtungen sollte jede Zahl etwa 200 Mal zugewiesen werden.
Training und Validierung des Modells
cv.fun <- function(this.fold, data){
train <- filter(data, my.folds != this.fold)
validate <- filter(data, my.folds == this.fold)
model <- lm(y ~ NLIN_1 + NLIN_2 + LIN_1 + LIN_2,
data = train)
pred <- predict(model, newdata = validate) %>% as.vector()
this.rmse <- RMSE(f = pred, o = validate$y)
return(this.rmse)
}Anschließend definieren wir „cv.fun“, das Herzstück unseres Kreuzvalidierungsverfahrens. Diese Funktion benötigt zwei Argumente: this.fold und data. Auf die Bedeutung von this.fold werde ich gleich zurückkommen, für den Moment setzen wir es einfach auf 1. Innerhalb der Funktion teilen wir die Daten in eine Trainings- und eine Validierungspartition auf, indem wir sie nach den Werten von my.folds und this.fold unterteilen: Jede Beobachtung mit einem zufällig zugewiesenen my.folds-Wert abweichend von 1 (also etwa 4/5 der Daten) geht in das Training. Alle Beobachtungen mit einem my.folds-Wert gleich 1 (die restlichen 1/5) bilden die Validierungsmenge. Zur Veranschaulichung passen wir dann ein einfaches lineares Modell mit dem simulierten Ergebnis und vier Prädiktoren an. Beachte, dass wir dieses Modell nur auf die train-Daten anwenden! Dann verwenden wir dieses Modell, um unsere Validierungsdaten vorherzusagen. Da wir für diese Teilmenge der ursprünglichen Trainingsdaten wahre Ergebnisse haben (darum geht es ja!), können wir unseren RMSE berechnen und ihn zurückgeben.
Iterieren durch die Foldings und Berechnen des CV-Fehlers
cv.error <- sapply(seq_len(k),
FUN = cv.fun,
data = sim_data) %>%
mean()
cv.errorZum Schluss verpacken wir den Funktionsaufruf von cv.fun in eine sapply()-Schleife – hier passiert die ganze Magie: Wir iterieren über den Bereich von k, so dass seq_len(k) uns in diesem Fall den Vektor [1] 1 2 3 4 5 liefert. Wir wenden jedes Element dieses Vektors auf cv.fun an. In der *apply()-Familie wird der Iterationsvektor immer als erstes Argument der aufgerufenen Funktion übergeben, so dass in unserem Fall jedes Element dieses Vektors zu einem Zeitpunkt an this.fold übergeben wird. Außerdem übermitteln wir unsere simulierten sim_data als Argument data.
Fassen wir kurz zusammen, was das bedeutet: In der ersten Iteration ist „this.fold“ gleich 1. Das bedeutet, dass unsere Trainingsmenge aus allen Beobachtungen besteht, bei denen my.folds nicht 1 ist, und die Beobachtungen mit dem Wert 1 bilden die Validierungsmenge (genau wie im Beispiel oben). In der nächsten Iteration der Schleife ist this.fold gleich 2. Folglich bilden die Beobachtungen mit den Werten 1, 3, 4 und 5 die Trainingsmenge und die Beobachtungen mit dem Wert 2 gehen in die Validierungsgruppe, und so weiter. Indem wir über alle Werte von k iterieren, erhalten wir das in Abbildung 2 gezeigte diagonale Muster, bei dem jede Datenpartition einmal als Validierungsmenge verwendet wird.
Zum Schluss berechnen wir den Mittelwert: Das ist der Mittelwert unserer k einzelnen RMSE-Werte und ergibt unseren Kreuzvalidierungsfehler. Und das war’s: Wir haben gerade unsere eigene Kreuzvalidierungsfunktion definiert!
Dies ist lediglich eine Vorlage: Du kannst jedes beliebige Modell und jede beliebige Fehlermetrik einfügen. Wenn du bis hierher durchgehalten hast, kannst du gerne selbst versuchen, eine wiederholte CV zu implementieren oder mit verschiedenen Werten für k herumzuspielen.
Fazit
Wie du siehst, ist es gar nicht so schwer, die Kreuzvalidierung selbst zu implementieren. Sie bietet dir eine große Flexibilität, um projektspezifische Anforderungen zu berücksichtigen, wie z. B. benutzerdefinierte Fehlermetriken. Wenn du nicht so viel Flexibilität brauchst, ist die Implementierung der Kreuzvalidierung in gängigen Machine Learning-Paketen ein Kinderspiel.
Ich hoffe, dass ich dir einen ausreichenden Überblick über die Kreuzvalidierung geben konnte und wie du sie sowohl in vordefinierten Funktionen als auch von Hand implementieren kannst. Wenn du Fragen, Kommentare oder Ideen hast, kannst du mir gerne eine E-Mail schicken.
Quellen
Nearly one year ago, I analyzed how we use emojis in our Slack messages. Since then, STATWORX grew, and we are a lot more people now! So, I just wanted to check if something changed.
Last time, I did not show our custom emojis, since they are, of course, not available in the fonts I used. This time, I will incorporate them with geom_image(). It is part of the ggimage package from Guangchuang Yu, which you can find here on his Github. With geom_image() you can include images like .png files to your ggplot.
What changed since last year?
Let’s first have a look at the amount of emojis we are using. In the plot below, you can see that since my last analysis in October 2018 (red line) the amount of emojis is rising. Not as much as I thought it would, but compared to the previous period, we now have more days with a usage of over 100 emojis per day!
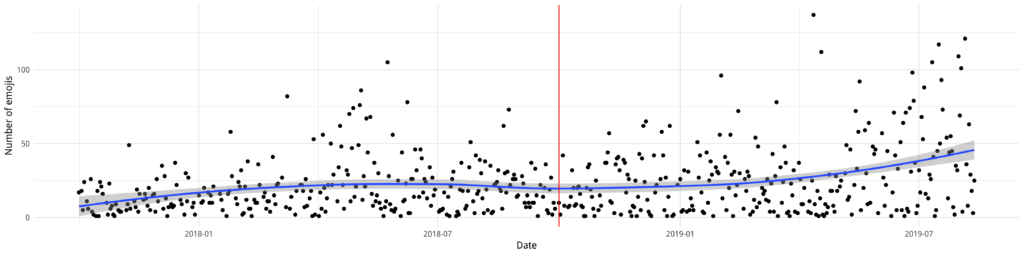
Like last time, our top emoji is ????, followed by ???? and ????. But sneaking in at number ten is one of our custom emojis: party_hat_parrot!
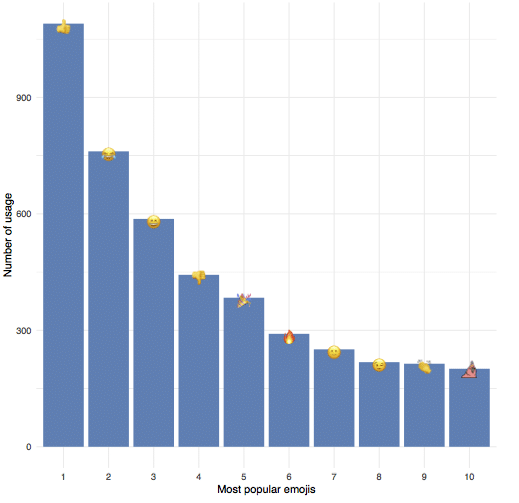
How to include custom images?
In my previous blogpost, I hid all our custom emojis behind❓since they were not part of the font. It did not occur to me to use their images, even though the package is from the same creator! So, to make up for my ignorance, I grabbed the top 30 custom emojis and downloaded their images from our Slack servers, saved them as .png and made sure they are all roughly the same size.
To use geom_image() I just added the path of the images to my data (the … are just an abbreviation for the complete path).
NAME COUNT REACTION IMAGE 1: alnatura 25 63 .../custom/alnatura.png 2: blog 19 20 .../custom/blog.png 3: dataiku 15 22 .../custom/dataiku.png 4: dealwithit_parrot 3 100 .../custom/dealwithit_parrot.png 5: deananddavid 31 18 .../custom/deananddavid.png
This would have been enough to just add the images now, but since I wanted the NAME attribute as a label, I included geom_text_repel from the ggrepel library. This makes handling of non-overlapping labels much simpler!
ggplot(custom_dt, aes( x = REACTION, y = COUNT, label = NAME)) +
geom_image(aes(image = IMAGE), size = 0.04) +
geom_text_repel(point.padding = 0.9, segment.alpha = 0) +
xlab("as reaction") +
ylab("within message") +
theme_minimal()
Usually, if a label is „too far“ away from the marker, geom_text_repel includes a line to indicate where the labels belong. Since these lines would overlap the images, I used segment.alpha = 0 to make them invisible. With point.padding = 0.9 I gave the labels a bit more space, so it looks nicer. Depending on the size of the plot, this needs to be adjusted. In the plot, one can see our usage of emojis within a message (y-axis) and as a reaction (x-axis).
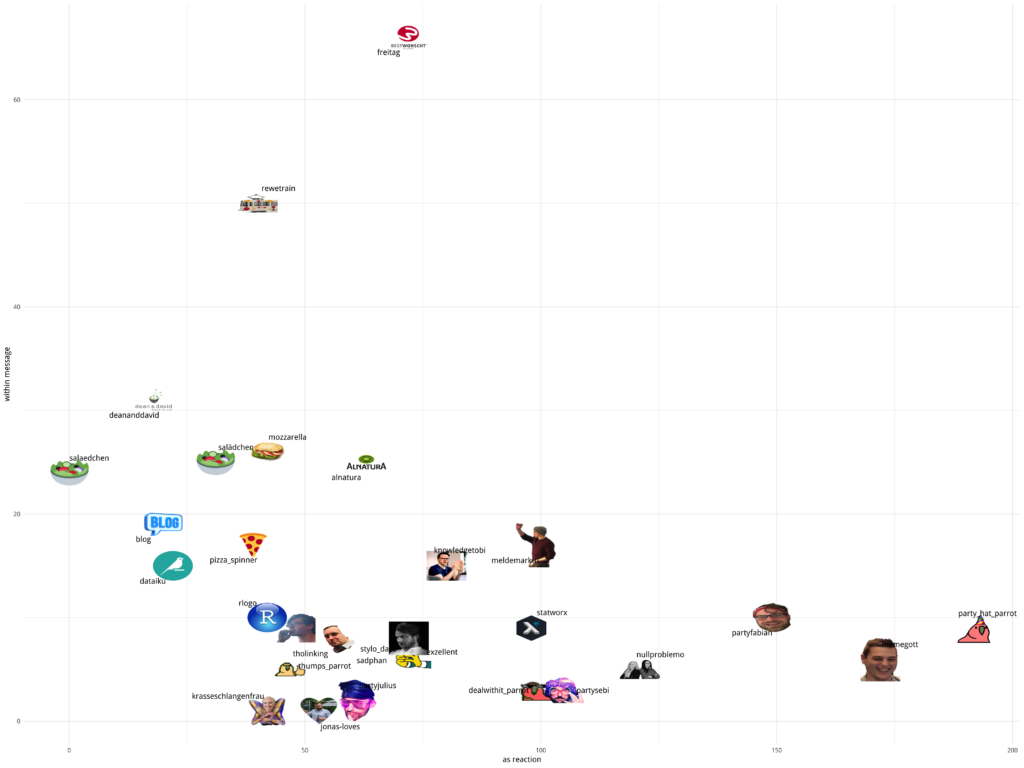
To combine the emoji font and custom emojis, I used the following data and code — really… why did I not do this last time? ???? Since the UNICODE is NA when I want to use the IMAGE, there is no „double plotting“.
EMOJI REACTION COUNT SUM PLACE UNICODE IMAGE 1: :+1: 1090 0 1090 1 U0001f44d 2: :joy: 609 152 761 2 U0001f602 3: :smile: 91 496 587 3 U0001f604 4: :-1: 434 9 443 4 U0001f44e 5: :tada: 346 38 384 5 U0001f389 6: :fire: 274 17 291 6 U0001f525 7: :slightly_smiling_face: 1 250 251 7 U0001f642 8: :wink: 27 191 218 8 U0001f609 9: :clap: 201 13 214 9 U0001f44f 10: :party_hat_parrot: 192 9 201 10 <NA> .../custom/party_hat_parrot.png
quartz()
ggplot(plotdata2, aes(x = PLACE, y = SUM, label = UNICODE)) +
geom_bar(stat = "identity", fill = "steelblue") +
geom_text(family="EmojiOne") +
xlab("Most popular emojis") +
ylab("Number of usage") +
scale_fill_brewer(palette = "Paired") +
geom_image(aes(image = IMAGE), size = 0.04) +
theme_minimal()
ps = grid.export(paste0(main_path, "plots/top-10-used-emojis.svg"), addClass=T)
dev.off()
The meaning behind emojis
Now we know what our top emojis are. But what is the rest of the world doing? Thanks to Emojimore for providing me with this overview! On their site, you can find meanings for a lot more emojis.
Behind each of our custom emojis is a story as well. For example, all the food emojis are helping us every day to decide where to eat and provide information on what everyone is planning for lunch! And if you do not agree with the decision, just react with sadphan to let the others know about your feelings. If you want to know the whole stories behind all custom emojis or even help create new ones, then maybe you should join our team — check out our available job offers here!
Mein Blogbeitrag zielt auf Data Science Einsteiger ab, die vor der Wahl stehen, welche Programmiersprache sie als Erstes lernen wollen. Wir bei statworx arbeiten mit den zwei beliebtesten Sprachen R und Python. Beide Sprachen haben ihre Stärken und Schwächen, weshalb man idealerweise beide beherrschen sollte. Für den Einstieg empfehlen wir eine Sprache zu erlernen und sich dann in der anderen fortzubilden. Um die Entscheidung zu erleichtern, mit welcher Programmiersprache man beginnen möchte, stelle ich Euch beide vor und vergleiche sie anschließend miteinander.
Überblick R und Python
Sowohl Python als auch R sind Open-Source-Programmiersprachen. Das bedeutet, dass die Quellcodes öffentlich zugänglich sind und gratis verwendet werden können. Während Python eine General Purpose Programmiersprache (Allzwecksprache) ist, wurde R für statistische Analysen entwickelt. Daher weisen die Nutzer der Sprachen oftmals unterschiedliche Hintergründe auf. Verallgemeinernd kann man sagen, dass Softwareentwickler Python nutzen und Statistiker R.
| R | Python | |
|---|---|---|
| Veröffentlichung | 1993 | 1991 |
| Entwickler | R Core Team | Python Software Foundation |
| Package Management | CRAN | Conda (empfohlen für Einsteiger) |
Eine Fülle an Erweiterungen
Beide Sprachen verfügen über einen Grundstock an Funktionen, die mit Paketen (packages) erweitert werden können.
Das Comprehensive R Archive Network (CRAN) ist eine Plattform für R Pakete. Um ein Paket auf CRAN bereit zu stellen, müssen eine ganze Reihe an Richtlinien eingehalten werden. CRAN gewährleistet dadurch, dass alle Pakete, die dort zum Download zur Verfügung stehen auch tatsächlich funktionieren. Insgesamt stehen auf CRAN 10.000 Pakete zur Verfügung. Da R die Standard-Sprache für Statistiker ist, findet man in CRAN für fast jedes Problem im Bereich Statistik eine passende Lösung. Es ist also genau die richtige Anlaufstelle für die neuesten statistischen Methoden und Analysen.
Bei Python gibt es zwei Paket-Verwaltungsplattformen: conda und PyPI (Python Package Index). Auch für Python gibt es über 10.000 Pakete, die im Gegensatz zu R einen sehr breiten Anwendungsbereich abdecken. Da es zu Komplikationen kommen kann, wenn Python Pakete global installiert werden, nutzt man dafür virtuelle Umgebungen. Die sorgen für reibungslose Abläufe innerhalb der verschiedenen Pakete und bei Abhängigkeiten von Paket zu Paket. Für Anfänger ist es daher nicht so einfach, sich da zurecht zu finden.
Mit Hilfe von Paketen besteht die Möglichkeit in R Python Code auszuführen sowie vice versa. Falls dich das interessiert, check den Blogbeitrag von meinem Kollegen Manuel ab. Er stellt das Paket reticulate vor.
IDEs als Hilfestellung
Programmierer nutzen oftmals eine integrierte Entwicklungsumgebung (IDE), die ihnen die Arbeit durch kleine aber feine Hilfsmittel erleichtert.
Für R Nutzer hat sich RStudio als Standard-IDE durchgesetzt. Die IDE wird vom gleichnamigen Unternehmen vertrieben, das kommerziell hinter R steht. RStudio bietet nicht nur ein angenehmes Arbeitsumfeld, sondern entwickelt auch aktiv Pakete und Erweiterungen für die R Sprache. Vom RStudio-Team stammen beispielsweise wichtige Pakete wie tidyverse, packrat und devtools sowie beliebte Erweiterungen wie shiny (Dashboards) und RMarkdown (Berichte).
Python Nutzer haben die Wahl zwischen verschiedenen IDEs (PyCharm, Visual Studio Code, Spyder, …). Allerdings gibt es kein Unternehmen, das hinter Python steht und vergleichbar mit RStudio wäre. Dennoch werden dank der Bemühungen der riesigen Community und der Python Software Foundation ständig neue Erweiterungen für Python zusammengestellt.
Die Kunst der Datenvisualisierung
Die meist verwendeten Pakete für Datenvisualisierung mit Python sind matplotlib und seaborn. Dashboards lassen sich in Python mit dash erstellen.
Aber R hat bei der Datenvisualisierung einen Trumpf im Ärmel: Das Paket ggplot2, das auf dem Buch The Grammar of Graphics von Leland Wilkinson basiert. Mit diesem Paket kannst Du ansprechende und maßgeschneiderte Grafiken erstellen, die Du wiederum auf Dashboards mit Hilfe von shiny für Andere zugänglich machen kannst.
Beide Programmiersprachen bieten die Möglichkeit, schöne Grafiken leicht zu erstellen. Trotzdem überzeugt das R Paket ggplot2 mit seiner Flexibilität und seinen visuellen Möglichkeiten.
Pluspunkte für Lesbarkeit
Python wurde nach dem Motto Readability counts konzipiert. Somit können auch Leute, die nicht mit der Programmiersprache vertraut sind, interpretieren was im Code gemacht wird.
Das ist in R Code eher nicht der Fall. Die Sprache ist weniger intuitiv aufgebaut als Python. Aufgrund der guten Lesbarkeit bietet Python daher einen leichteren Einstieg ins Programmieren.
Schnelligkeit in verschiedenen Observationsgrößen
Als nächstes vergleiche ich, wie lange es dauert in R und Python einen simulierten Datensatz zu erstellen. Für eine faire Gegenüberstellung sollten die Bedingungen möglichst gleich sein. Die Daten werden mit den Paketen Xy und XyPy in R und Python respektive simuliert. Für die Zeitmessung habe ich microbenchmark in R und timeit in Python benutzt. Um die Simulation schnellstmöglich zu generieren, wird der Prozess parallelisiert auf acht Kernen (R: parallel, Python: multiprocessing).
Für das Experiment wird ein Datensatz mit 100 Observationen und 50 Variablen 100 Mal simuliert. Die Zeit, die der Rechner benötigt, um die Simulation durchzuführen, wird für jede Simulation einzeln gemessen. Und das wird dann für 1.000, 10.000, 100.000 und 1.000.000 Observationen wiederholt.
Die R und Python Code Snippets sind unten abgebildet.
# R
# devtools::install_github("andrebleier/Xy")
# install.packages("parallel")
# install.packages("microbenchmark")
# Load packages
library(Xy)
library(microbenchmark)
library(parallel)
# Extract function definition from for loop
sim_this <- function(n_sim) {
sim <- microbenchmark(Xy(n = n_sim,
numvars = c(50,0),
catvars = 0),
times = 100, unit = "s")
data.frame(n = n_sim,
mean = summary(sim)[, 4])
}
# Time measurement for different number of simulations
n_sim <- c(1e2, 1e3, 1e4, 1e5, 1e6)
sim_in_r <- data.frame(n = rep(0, length(n_sim)),
t = rep(0, length(n_sim)))
for(i in 1:length(n_sim)){
out <- mclapply(n_sim[i],
FUN = sim_this,
mc.cores = 8)
sim_in_r[i, 1] <- out[[1]][1]
sim_in_r[i, 2] <- out[[1]][2]
}
# Python
# In terminal: pip install xypy
import multiprocessing as mp
import numpy as np
import timeit
from xypy import Xy
# Predefine function of interest
def sim_this(n_sim):
return(timeit.timeit( lambda: Xy(n = int(n_sim),
numvars = [50, 0],
catvars = [0, 0],
weights = [5, 10],
stn = 4.0,
cor = [0, 0.1],
interactions = 1,
noisevars = 5), number = 100))
# Paralleled computation
pool = mp.Pool(processes = 8)
n_sim = np.array([1e2, 1e3, 1e4, 1e5, 1e6])
results = [pool.map(sim_this, n_sim)]
Die durchschnittliche Dauer, sortiert nach Datensatzgröße, wird für R und Python im unteren Plot dargestellt. Die X-Achse wird hier auf einer logarithmischen Skala mit Basis 10 dargestellt, um die Grafik übersichtlicher zu machen.
Während R bei einer Datensatzgröße von 100 und 1.000 Observationen etwas schneller ist, hängt Python R bald darauf deutlich ab.
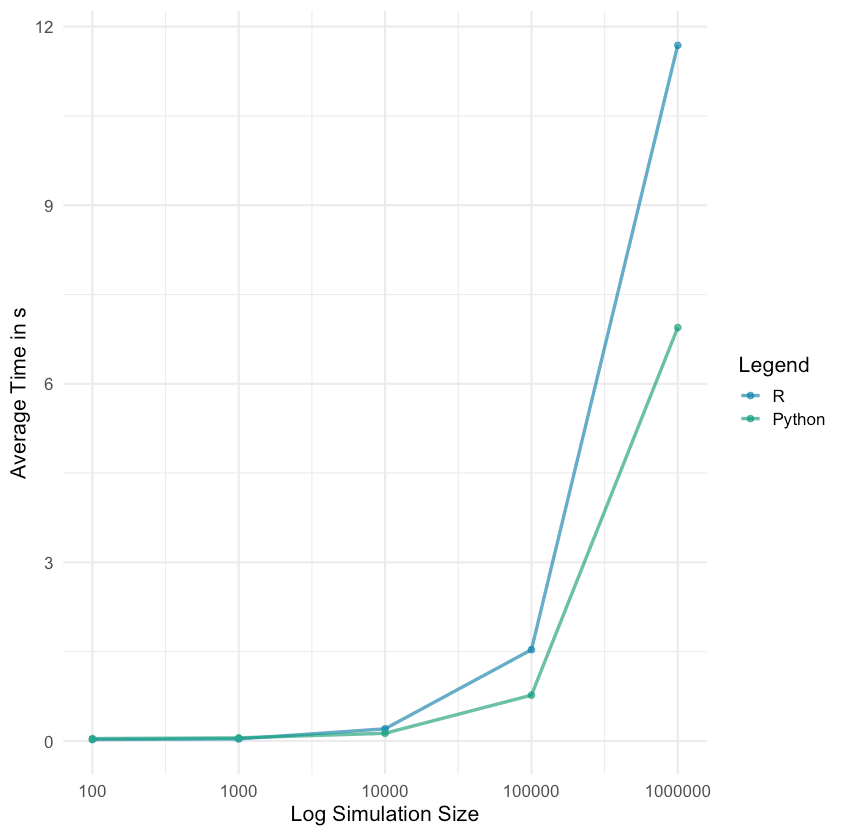
Für weitere Vergleiche kann ich die folgenden STATWORX Blogbeiträge empfehlen: pandas vs. data.table und pandas vs. data.table part 2, dabei wird der Fokus auf Datenmanipulation gelegt.
Der Standard bei Deep Learning
Wenn Dich vor allem Deep Learning Methoden interessieren, eignet sich Python als Sprache besser. Die meisten Deep Learning Bibliotheken wurden in Python geschrieben und implementiert.
Auch in R ist Deep Learning möglich, aber die R Deep Learning Community ist deutlich kleiner. Implementationen wie Keras und TensorFlow lassen sich zwar auch in R aufrufen, dies läuft dann aber über Pakete von Drittanbietern. Die Pakete bieten daher nicht die volle Flexibilität für die Nutzer, z.B. sind nicht alle TensorFlow Funktionen erhältlich. Zu dem kommt der Aspekt der Schnelligkeit. Deep Learning mit Python ist schneller als mit R.
Umfrage in der Community: Wie ticken die Anwender?
Als angehende Data Scientists ist Kaggle eine wichtige Plattform für Euch. Dort kann man an spannenden Machine Learning Wettkämpfen teilnehmen, selbst experimentieren und aus den Erfahrungen der Community lernen.
2018 hat Kaggle eine Machine Learning & Data Science Umfrage durchgeführt. Die Umfrage war zwei Wochen lang online und es gingen insgesamt 23.859 Antworten ein. Aus den Ergebnissen dieser Umfrage habe ich verschiedene Plots erstellt, aus denen sich einige interessante Schlüsse im Hinblick auf mein Blogthema ziehen lassen. Der Code zu den einzelnen Plots ist öffentlich zugänglich auf Github.
Exkurs: Python & R im Vergleich zu anderen Sprachen
Bevor wir uns auf R und Python stürzen, schauen wir uns an, wie die beiden im Vergleich zu anderen Programmiersprachen abschneiden. Jeder Umfragenteilnehmer gab an, welche Sprache er vorrangig benutzt. Im unteren Plot wurde nach Sprache aggregiert und das Ergebnis lautet: Die große Mehrzahl der Teilnehmer benutzt vor allem Python! Gefolgt von R auf dem zweiten Platz. In dieser Umfrage unterscheiden wir nicht zwischen den Arbeitsbereichen, weshalb Python – als General Purpose Programmiersprache – vermutlich so stark hervorsticht.
Die Gegenüberstellung R & Python
Im direkten Vergleich zwischen R und Python sieht man, dass sehr viele R-Nutzer auch Python benutzen. Wohingegen die Python-Nutzer oftmals ausschließlich mit Python arbeiten.
Wenn man die Nutzung der Sprachen nach Arbeitsbereich vergleicht, sieht man eine klare Dominanz von Python. In allen Arbeitsfeldern, bis auf Statistiker, wird mehrheitlich Python benutzt.

Die Teilnehmer wurden außerdem gefragt: Welche Sprache empfiehlst Du angehenden Data Scientists zuerst zu lernen? Die Antworten auf die Frage sind in der unteren Tabelle zusammengefasst.
| Sprache | Empfehlung | Nutzer | Differenz |
|---|---|---|---|
| Python | 14.181 | 8.180 | 6.001 |
| R | 2.342 | 2.046 | 296 |
| SQL | 914 | 1.211 | -297 |
| C++ | 339 | 739 | -400 |
| Matlab | 256 | 355 | -99 |
| Java | 184 | 903 | -719 |
| Scala | 74 | 106 | -32 |
| Javascript | 72 | 408 | -336 |
| SAS | 69 | 228 | -159 |
| VBA | 38 | 135 | -97 |
| Go | 26 | 46 | -20 |
| Other | 161 | 117 | 44 |
Wenn man die Anzahl Empfehlungen und die Anzahl Nutzer vergleicht, dann sieht man, dass R und Python die einzigen Sprachen sind, die eine positive Differenz aufweisen.
Auch bei dieser Frage liegt Python (14.181) wieder weit vor R (2.342).
Fazit
Eine Sache vorweg: beide Sprachen sind sehr mächtig. Daher kann man keine falsche Wahl treffen! Die Wahl der Sprache hängt davon ab, welche Projekte man verwirklichen möchte.
Als universelle Programmiersprache ist Python für diverse Anwendungsgebiete geeignet. Weshalb ich Dir grundsätzlich empfehle mit Python anzufangen. Falls aber statistische Auswertungen oder Datenvisualisierungen bei Deinen Projekten im Vordergrund stehen, hat R gegenüber Python einen Vorteil.
Wie schon erwähnt haben beide Sprachen ihre Vor- und Nachteile. Als fortgeschrittener Data Scientist solltest Du idealerweise beide Sprachen beherrschen.
Ich hoffe, dass Dir dieser Beitrag bei der Suche nach dem richtigen Einstieg in die Data Science Welt weiterhilft.
Happy Coding!
Falls Du Interesse an Schulungen hast, kannst du dir gerne unter AI Academy unsere Kurse durchschauen.
Referenzen
- https://cran.r-project.org/
- https://www.python.org/
- https://wiki.c2.com/?PythonPhilosophy
- https://plot.ly/
- https://www.rstudio.com/
- https://pipenv.readthedocs.io/en/latest/
- https://conda.io/en/latest/
- https://www.kaggle.com
- https://www.springer.com/us/book/9780387245447
- https://www.statworx.com/ai-academy/
Der Vergleich von Prognosemethoden und Modellen über verschiedene Zeitreihen hinweg ist oft problematisch. Dieser Herausforderung sehen wir uns bei STATWORX regelmäßig gegenüber. Einheitenabhängige Maße wie der MAE (Mean Absolute Error) und der RMSE (Root Mean Squared Error) erweisen sich als ungeeignet und wenig hilfreich, wenn die Zeitreihen in unterschiedlichen Einheiten gemessen werden. Ist dies jedoch nicht der Fall, liefern beide Maße wertvolle Informationen. Der MAE ist gut interpretierbar, da er die durchschnittliche absolute Abweichung von den tatsächlichen Werten angibt. Der RMSE hingegen ist nicht so einfach zu interpretieren und anfälliger für Extremwerte, wird aber in der Praxis dennoch häufig verwendet.
![]()
![]()
Eine der am häufigsten verwendeten Messgrößen, die dieses Problem vermeidet, heißt MAPE (Mean Absolute Percentage Error). Er löst das Problem der genannten Ansätze, da er nicht von der Einheit der Zeitreihe abhängt. Außerdem können Entscheidungsträger ohne statistisches Hintergrundwissen dieses Maß leicht interpretieren und verstehen. Trotz seiner Beliebtheit wurde und wird der MAPE immer noch kritisiert.
![]()
In diesem Artikel bewerte ich diese kritischen Argumente und zeige, dass zumindest einige von ihnen höchst fragwürdig sind. Im zweiten Teil meines Artikels konzentriere ich mich auf die wahren Schwächen des MAPE. Im dritten Teil diskutiere ich verschiedene Alternativen und fasse zusammen, unter welchen Umständen die Verwendung des MAPE sinnvoll erscheint (und wann nicht).
Was dem MAPE alles FÄLSCHLICH vorgeworfen wird
Negative Fehler werden stärker bestraft als positive Fehler
Die meisten Quellen, die sich mit dem MAPE beschäftigen, weisen auf dieses „große“ Problem der Messung hin. Diese Aussage basiert hauptsächlich auf zwei verschiedenen Argumenten. Erstens wird behauptet, dass der Austausch des tatsächlichen Wertes mit dem prognostizierten Wert die Richtigkeit der Aussage beweist (Makridakis 1993).
Fall 1: ![]() = 150 &
= 150 & ![]() = 100 (positive error)
= 100 (positive error)
![]()
Fall 2: ![]() = 100 &
= 100 & ![]() = 150 (negative error)
= 150 (negative error)
![]()
Es stimmt, dass Fall 1 (positiver Fehler von 50) mit einem niedrigeren APE (Absolute Percentage Error) verbunden ist als Fall 2 (negativer Fehler von 50). Der Grund dafür ist jedoch nicht, dass der Fehler positiv oder negativ ist, sondern einfach, dass sich der tatsächliche Wert ändert. Wenn der tatsächliche Wert konstant bleibt, ist der APE für beide Fehlerarten gleich *(Goodwin & Lawton 1999)*. Das wird durch das folgende Beispiel verdeutlicht.
Fall 3: ![]() = 100 &
= 100 & ![]() = 50
= 50
![]()
Fall 4: ![]() = 100 &
= 100 & ![]() = 150
= 150
![]()
The second, equally invalid argument supporting the asymmetry of the MAPE arises from the assumption about the predicted data. As the MAPE is mainly suited to be used to evaluate predictions on a ratio scale, the MAPE is bounded on the lower side by an error of 100% (Armstrong & Collopy 1992). However, this does not imply that the MAPE overweights or underweights some types of errors, but that these errors are not possible.
Seine WAHREN Schwächen
Tatsächliche Werte gleich Null sind problematisch
Diese Aussage ist ein bekanntes Problem des Maßes. Dieses und das letztgenannte Argument waren der Grund für die Entwicklung einer modifizierten Form des MAPE, des SMAPE („Symmetric“ Mean Absolute Percentage). Ironischerweise leidet diese modifizierte Form im Gegensatz zum ursprünglichen MAPE unter einer echten Asymmetrie (Goodwin & Lawton 1999). Ich werde dieses Argument im letzten Abschnitt des Artikels erläutern.
Besonders kleine tatsächliche Werte verzerren den MAPE
Wenn ein wahrer Wert sehr nahe bei Null liegt, sind die entsprechenden absoluten prozentualen Fehler extrem hoch und verzerren daher die Aussagekraft des MAPE (Hyndman & Koehler 2006). Die folgende Grafik verdeutlicht diesen Punkt. Obwohl alle drei Prognosen die gleichen absoluten Fehler aufweisen, ist der MAPE der Zeitreihe mit nur einem extrem kleinen Wert etwa doppelt so hoch wie der MAPE der anderen Prognosen. Dieser Sachverhalt impliziert, dass der MAPE bei extrem kleinen Beobachtungen vorsichtig verwendet werden sollte und motiviert direkt die letzte und oft ignorierte Schwäche des MAPE.
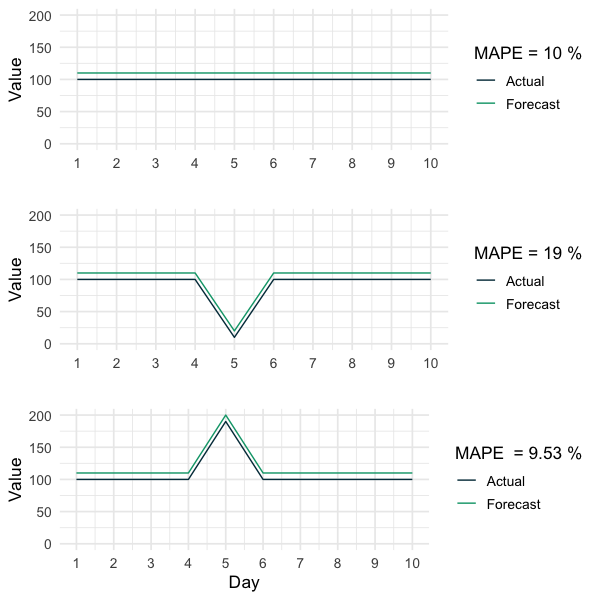
Der MAPE sagt nur aus, welche Prognose verhältnismäßig besser ist
Wie bereits zu Beginn dieses Artikels erwähnt, liegt ein Vorteil der Verwendung des MAPE für den Vergleich zwischen Prognosen verschiedener Zeitreihen in seiner Unabhängigkeit von der Einheit. Es ist jedoch wichtig zu bedenken, dass der MAPE nur angibt, welche Prognose proportional besser ist. Die folgende Grafik zeigt drei verschiedene Zeitreihen und die dazugehörigen Prognosen. Der einzige Unterschied zwischen ihnen ist ihr allgemeines Niveau. Dieselben absoluten Fehler führen also zu völlig unterschiedlichen MAPEs. In diesem Artikel wird kritisch hinterfragt, ob es sinnvoll ist, ein solches prozentuales Maß für den Vergleich zwischen Prognosen für verschiedene Zeitreihen zu verwenden. Wenn sich die verschiedenen Zeitreihen nicht auf einer irgendwie vergleichbaren Ebene verhalten (wie in der folgenden Grafik dargestellt), beruht die Verwendung des MAPE, um zu ermitteln, ob eine Prognose für eine Zeitreihe allgemein besser ist als für eine andere, auf der Annahme, dass dieselben absoluten Fehler für Zeitreihen auf höheren Ebenen weniger problematisch sind als für Zeitreihen auf niedrigeren Ebenen:
„Wenn eine Zeitreihe um 100 schwankt, dann ist die Vorhersage von 101 viel besser als die Vorhersage von 2 für eine Zeitreihe, die um 1 schwankt.“
Das mag in manchen Fällen stimmen. Im Allgemeinen ist dies jedoch eine Annahme, der man sich immer bewusst sein sollte, wenn man den MAPE-Wert zum Vergleich von Prognosen zwischen verschiedenen Zeitreihen verwendet.
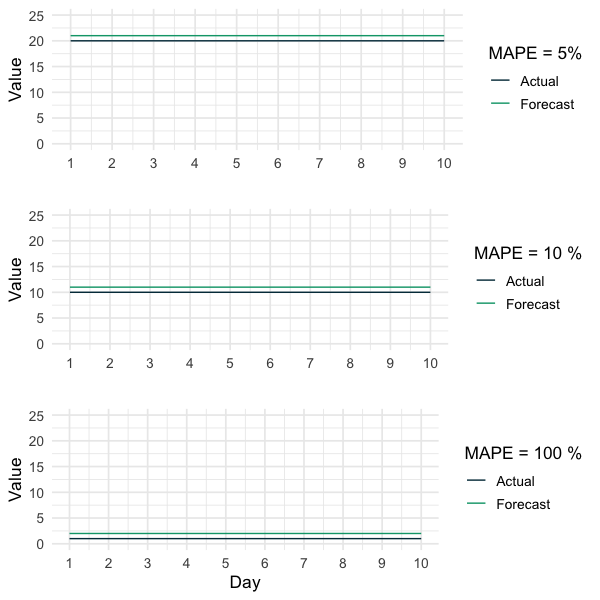
Zusammenfassung
Insgesamt zeigen die diskutierten Ergebnisse, dass der MAPE mit Vorsicht als Instrument für den Vergleich von Prognosen zwischen verschiedenen Zeitreihen verwendet werden sollte. Eine notwendige Bedingung ist, dass die Zeitreihe nur streng positive Werte enthält. Zweitens haben nur einige extrem kleine Werte das Potenzial, den MAPE stark zu verzerren. Und schließlich hängt der MAPE systematisch von dem Level der Zeitreihe ab, da es sich um einen prozentualen Fehler handelt. In diesem Artikel wird kritisch hinterfragt, ob es sinnvoll ist, von einer proportional besseren Prognose auf eine allgemein bessere Prognose zu verallgemeinern.
Bessere Alternativen
Die Diskussion zeigt, dass der MAPE allein oft nicht sehr nützlich ist, wenn es darum geht, die Genauigkeit verschiedener Prognosen für unterschiedliche Zeitreihen zu vergleichen. Obwohl es bequem sein kann, sich auf ein leicht verständliches Maß zu verlassen, birgt es ein hohes Risiko für irreführende Schlussfolgerungen. Im Allgemeinen ist es immer empfehlenswert, verschiedene Messwerte kombiniert zu verwenden. Zusätzlich zu den numerischen Messwerten liefert eine Visualisierung der Zeitreihe, einschließlich der tatsächlichen und der prognostizierten Werte, immer wertvolle Informationen. Wenn ein einzelnes numerisches Maß jedoch die einzige Option ist, gibt es einige erwähnenswerte Alternativen.
Scaled Measures
Scaled Measures vergleichen das Maß einer Prognose, z.B. den MAE im Verhältnis zum MAE einer Benchmark-Methode. Ähnliche Maße können mit RMSE, MAPE oder anderen Maßen definiert werden. Gängige Benchmark-Methoden sind der „random walk“, die „naive“ Methode und die „mean“ Methode. Diese Scaled Measures sind leicht zu interpretieren, da sie zeigen, wie das Schwerpunktmodell im Vergleich zu den Benchmark-Methoden abschneidet. Es ist jedoch wichtig zu bedenken, dass sie von der Auswahl der Benchmark-Methode abhängen und davon, wie gut die Zeitreihe mit der gewählten Methode vorhergesagt werden kann.
![]()
Scales Errors
Scaled Errors versuchen ebenfalls, die Skalierung der Daten zu beseitigen, indem sie die prognostizierten Werte mit denen einer Benchmark-Prognosemethode, wie der naiven Methode, vergleichen. Der MASE (Mean Absolute Scaled Error), der von *Hydnmann & Koehler 2006* vorgeschlagen wurde, ist je nach Saisonalität der Zeitreihe leicht unterschiedlich definiert. Im einfachen Fall einer nicht saisonalen Zeitreihe wird der Fehler der Prognose auf der Grundlage des In-Sample-MASE der naiven Prognosemethode skaliert. Ein großer Vorteil ist, dass sie mit tatsächlichen Werten von Null umgehen kann und durch sehr extreme Werte nicht verzerrt wird. Auch hier ist zu beachten, dass die relativen Maße von der Auswahl der Benchmark-Methode abhängen und davon, wie gut die Zeitreihe mit der gewählten Methode vorhergesagt werden kann.
Nicht-Saisonal![]()
Saisonal![]()
SDMAE
Der Grundgedanke bei der Verwendung des MAPE zum Vergleich verschiedener Zeitreihen zwischen Prognosen ist meines Erachtens, dass derselbe absolute Fehler für Zeitreihen auf höheren Ebenen weniger problematisch ist als für Zeitreihen auf niedrigeren Ebenen. Anhand der zuvor gezeigten Beispiele denke ich, dass diese Idee zumindest fragwürdig ist.
Ich vertrete die Auffassung, dass die Bewertung eines bestimmten absoluten Fehlers nicht vom allgemeinen Niveau der Zeitreihe abhängen sollte, sondern von ihrer Variation. Dementsprechend ist das folgende Maß, der SDMAE (Standard Deviation adjusted Mean Absolute Error), ein Produkt der diskutierten Fragen und Überlegungen. Es kann zur Bewertung von Prognosen für Zeitreihen mit negativen Werten verwendet werden und leidet nicht darunter, dass die tatsächlichen Werte gleich Null oder besonders klein sind. Beachte, dass dieses Maß nicht für Zeitreihen definiert ist, die überhaupt nicht schwanken. Außerdem könnte es weitere Einschränkungen dieses Maßes geben, die mir derzeit nicht bekannt sind.
![]()
Zusammenfassung
Ich schlage vor, eine Kombination verschiedener Messgrößen zu verwenden, um ein umfassendes Bild von der Leistung der verschiedenen Prognosen zu erhalten. Ich empfehle außerdem, den MAPE durch eine Visualisierung der Zeitreihe, die die tatsächlichen und die prognostizierten Werte enthält, den MAE und einen Scaled Measure oder Scaled Error zu ergänzen. Der SDMAE sollte als alternativer Ansatz gesehen werden, der bisher noch nicht von einem breiteren Publikum diskutiert wurde. Ich bin dankbar für deine kritischen Gedanken und Kommentare zu dieser Idee.
Schlechtere Alternativen
SMAPE
Der SMAPE wurde geschaffen, um die Probleme des MAPE zu lösen. Damit wurde jedoch weder das Problem der extrem kleinen Ist-Werte noch die Niveauabhängigkeit des MAPE gelöst. Der Grund dafür ist, dass extrem kleine tatsächliche Werte in der Regel mit extrem kleinen Vorhersagen verbunden sind *(Hyndman & Koehler 2006)*. Außerdem wirft der SMAPE im Gegensatz zum unveränderten MAPE das Problem der Asymmetrie auf *(Goodwin & Lawton 1999)*. Dies wird durch die folgende Grafik verdeutlicht, wobei sich der „APE“ auf den MAPE und der „SAPE“ auf den SMAPE bezieht. Sie zeigt, dass der SAPE bei positiven Fehlern höher ist als bei negativen Fehlern und daher asymmetrisch. Die SMAPE wird von einigen Wissenschaftlern nicht empfohlen (Hyndman & Koehler 2006).
![]()
On the asymmetry of the symmetric MAPE_ _(Goodwin & Lawton 1999)
Quellen
- Goodwin, P., & Lawton, R. (1999). On the asymmetry of the symmetric MAPE. *International journal of forecasting*, *15*(4), 405-408.
Hyndman, R. J., & Koehler, A. B. (2006). Another look at measures of forecast accuracy. *International journal of forecasting*, *22*(4), 679-688.
Makridakis, S. (1993). Accuracy measures: theoretical and practical concerns. *International Journal of Forecasting*, *9*(4), 527-529.
Armstrong, J. S., & Collopy, F. (1992). Error measures for generalizing about forecasting methods: Empirical comparisons. *International journal of forecasting*, *8*(1), 69-80.
Netzwerke sind überall. Wir haben soziale Netzwerke wie Facebook, kompetitive Produktnetzwerke oder verschiedene Netzwerke innerhalb einer Organisation. Auch für STATWORX ist es eine häufige Aufgabe, verborgene Strukturen und Cluster in einem Netzwerk aufzudecken und sie für unsere Kunden zu visualisieren. In der Vergangenheit haben wir dazu das Tool Gephi verwendet, um unsere Ergebnisse der Netzwerkanalyse zu visualisieren. Beeindruckt von dieser schönen und interaktiven Visualisierung war unsere Idee, einen Weg zu finden, Visualisierungen in der gleichen Qualität direkt in R zu erstellen und sie unseren Kunden in einer R Shiny App zu präsentieren.
Unsere erste Überlegung war, Netzwerke mit igraph zu visualisieren, einem Paket, das eine Sammlung von Netzwerkanalysetools enthält, wobei der Schwerpunkt auf Effizienz, Portabilität und Benutzerfreundlichkeit liegt. Wir haben es in der Vergangenheit schon in unserem Paket helfRlein für die Funktion getnetwork verwendet. Das Paket igraph kann zwar ansprechende Netzwerkvisualisierungen erstellen, aber leider sind diese jedoch ausschließlich statisch. Um hingegen interaktive Netzwerkvisualisierungen zu erstellen, stehen uns mehrere Pakete in R zur Verfügung, die alle auf Javascript-Bibliotheken basieren.
Unser Favorit für diese Visualisierungsaufgabe ist das Paket visNetwork, das die Javascript-Bibliothek vis.js verwendet und auf htmlwidgets basiert. Es ist mit Shiny, R Markdown-Dokumenten und dem RStudio-Viewer kompatibel. visNetwork bietet viele Anpassungsmöglichkeiten, um ein Netzwerk zu individualisieren, einen schönen Output und eine gute Performance, was sehr wichtig ist, wenn wir den Output in Shiny verwenden möchten. Außerdem ist eine hervorragende Dokumentation hier zu finden.
Gehen wir nun also die Schritte durch, die von der Datenbasis bis hin zur perfekten Visualisierung in R Shiny gemacht werden müssen. Dazu verwenden wir im Folgenden das Netzwerk der Les Misérables Charaktere als Beispiel. Dieses ungerichtete Netzwerk enthält Informationen dazu, welche Figuren in Victor Hugos Roman „Les Misérables“ gemeinsam auftreten. Ein Knoten (engl. Node) steht für eine Figur, und ein Kante (engl. Edge) zwischen zwei Knoten zeigt an, dass diese beiden Figuren im selben Kapitel des Buches auftreten. Das Gewicht jeder Verbindung gibt an, wie oft ein solches gemeinsames Auftreten vorkommt.
Datenaufbereitung
Als erstes müssen wir das Paket mit install.packages("visNetwork") installieren und den Datensatz lesmis laden. Der Datensatz ist im Paket geomnet enthalten.
Um das Netzwerk zwischen den Charakteren von Les Misérables zu visualisieren, benötigt das Paket visNetwork zwei Datensätze. Einen für die Knoten und einen für die Kanten des Netzwerks. Glücklicherweise liefern unsere geladenen Daten beides und wir müssen sie nur noch in das richtige Format bringen.
rm(list = ls())
# Libraries ---------------------------------------------------------------
library(visNetwork)
library(geomnet)
library(igraph)
# Data Preparation --------------------------------------------------------
#Load dataset
data(lesmis)
#Nodes
nodes <- as.data.frame(lesmis[2])
colnames(nodes) <- c("id", "label")
#id has to be the same like from and to columns in edges
nodes$id <- nodes$label
#Edges
edges <- as.data.frame(lesmis[1])
colnames(edges) <- c("from", "to", "width")
Die folgende Funktion benötigt spezifische Namen für die Spalten, um die richtige Spalte zu erkennen. Zu diesem Zweck müssen die Kanten ein Data Frame mit mindestens einer Spalte sein, die angibt, in welchem Knoten eine Kante beginnt (from) und wo sie endet (to). Für die Knoten benötigen wir mindestens eine eindeutige ID (id), die mit den Argumenten from und to übereinstimmen muss.
Knoten:
label: Eine Spalte, die definiert, wie ein Knoten beschriftet istvalue: Definiert die Größe eines Knotens innerhalb des Netzwerksgroup: Weist einen Knoten einer Gruppe zu; dies kann das Ergebnis einer Clusteranalyse oder einer Community Detection seinshape: Legt fest, wie ein Knoten dargestellt wird. Zum Beispiel als Kreis, Quadrat oder Dreieckcolor: Legt die Farbe eines Knotens festtitle: Legt den Tooltip fest, der erscheint, wenn du den Mauszeiger über einen Knoten bewegst (dies kann HTML oder ein Zeichen sein)shadow: Legt fest, ob ein Knoten einen Schatten hat oder nicht (TRUE/FALSE)
Kanten:
label,title,shadowlength,width: Definiert die Länge/Breite einer Kante innerhalb des Netzwerksarrows: Legt fest, wo ein möglicher Pfeil an der Kante gesetzt werden solldashes: Legt fest, ob die Kanten gestrichelt sein sollen oder nicht (TRUE/FALSE)smooth: Darstellung als Kurve (TRUE/FALSE)
Dies sind die wichtigsten Einstellungen. Sie können für jeden einzelnen Knoten oder jede einzelne Kante individuell angepasst werden. Um einige Konfigurationen für alle Knoten oder Kanten einheitlich einzustellen, wie z.B. die gleiche Form oder Pfeile, können wir später bei der Spezifizierung des Outputs visNodes und visEdges nutzen. Wir werden diese Möglichkeit später noch genauer betrachten.
Zusätzlich wollen wir Gruppen innerhalb des Netzwerks identifizieren und darstellen. Wir werden dazu die Gruppen hervorheben, indem wir den Kanten des Netzwerks Farben hinzufügen. Dafür clustern wir die Daten zuerst mit der Community Detection Methode Louvain und erhalten die neue Spalte group:
#Create graph for Louvain
graph <- graph_from_data_frame(edges, directed = FALSE)
#Louvain Comunity Detection
cluster <- cluster_louvain(graph)
cluster_df <- data.frame(as.list(membership(cluster)))
cluster_df <- as.data.frame(t(cluster_df))
cluster_df$label <- rownames(cluster_df)
#Create group column
nodes <- left_join(nodes, cluster_df, by = "label")
colnames(nodes)[3] <- "group"
Output Optionen
Um einen Eindruck davon zu geben, welche Möglichkeiten wir haben, wenn es um die Design- und Funktionsoptionen bei der Erstellung unseres Output geht, sehen wir uns zwei Darstellungen des Les Misérables-Netzwerks genauer an.
Wir beginnen mit der einfachsten Möglichkeit und geben nur die Knoten und Kanten als Data Frames an die Funktion weiter:
visNetwork(nodes, edges)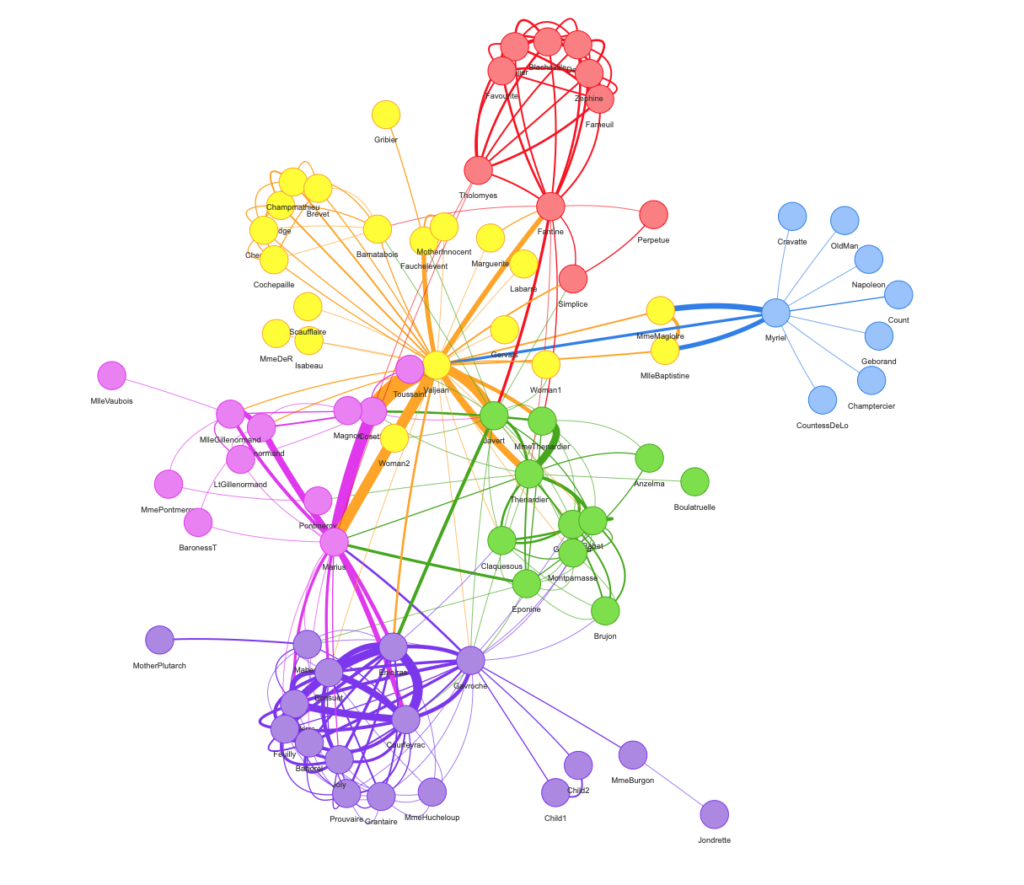
Mit dem Pipe-Operator können wir unser Netzwerk mit einigen anderen Funktionen wie visNodes, visEdges, visOptions, visLayout oder visIgraphLayout anpassen:
visNetwork(nodes, edges, width = "100%") %>%
visIgraphLayout() %>%
visNodes(
shape = "dot",
color = list(
background = "#0085AF",
border = "#013848",
highlight = "#FF8000"
),
shadow = list(enabled = TRUE, size = 10)
) %>%
visEdges(
shadow = FALSE,
color = list(color = "#0085AF", highlight = "#C62F4B")
) %>%
visOptions(highlightNearest = list(enabled = T, degree = 1, hover = T),
selectedBy = "group") %>%
visLayout(randomSeed = 11)
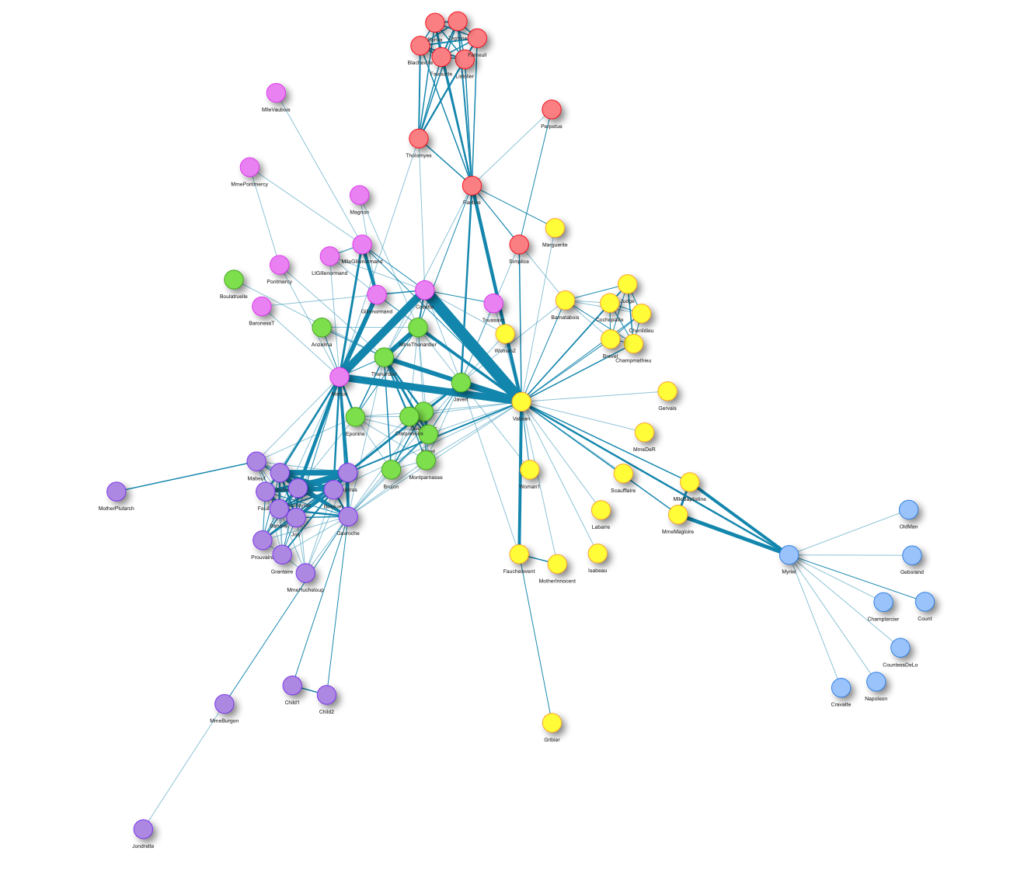
visNodes und visEdges beschreiben das allgemeine Aussehen der Knoten und Kanten im Netzwerk. Wir können zum Beispiel die Form aller Knoten festlegen oder die Farben aller Kanten definieren.
Wenn es um die Veröffentlichung in R geht, kann das Rendern des Netzwerks viel Zeit in Anspruch nehmen. Um dieses Problem zu lösen, verwenden wir die Funktion visIgraph. Sie verkürzt die Darstellungszeit, indem sie die Koordinaten im voraus berechnet und alle verfügbaren igraph-Layouts bereitstellt.
Mit visOptions können wir einstellen, wie das Netzwerk reagiert, wenn wir mit ihm interagieren. Zum Beispiel, was passiert, wenn wir auf einen Knoten klicken.
Mit visLayout können wir das Aussehen des Netzwerks festlegen. Soll es ein hierarchisches sein oder wollen wir das Layout mit einem speziellen Algorithmus verbessern? Außerdem können wir einen Seed (randomSeed) angeben, damit das Netzwerk immer identisch aussieht, wenn wir es laden.
Dies sind nur einige Beispielfunktionen, mit denen wir unser Netzwerk anpassen können. Das Paket bietet noch viel mehr Möglichkeiten zur individuellen Anpassung. Weitere Details sind in der Dokumentation zu finden.
Shiny Integration
Um die interaktiven Ergebnisse unseren Kunden zu präsentieren, wollen wir sie in eine Shiny-App integrieren. Deshalb bereiten wir die Daten „offline“ auf, speichern die Knoten- und Kanten-Dateien und erstellen den Output innerhalb der Shiny-App „online“. Hier ist ein einfaches Beispiel für das Schema, das für Shiny verwendet werden kann:
global.R:
library(shiny)
library(visNetwork)
server.R:
shinyServer(function(input, output) {
output$network <- renderVisNetwork({
load("nodes.RData")
load("edges.RData")
visNetwork(nodes, edges) %>%
visIgraphLayout()
})
})
ui.R:
shinyUI(
fluidPage(
visNetworkOutput("network")
)
)
Der Screenshot unserer Shiny-App veranschaulicht ein mögliches Ergebnis:
Fazit
Neben anderen verfügbaren Paketen zur interaktiven Visualisierung von Netzwerken in R, ist visNetwork unser absoluter Favorit. Es ist ein leistungsstarkes Paket, um interaktive Netzwerke direkt in R zu erstellen und in Shiny zu veröffentlichen. Wir können unsere Netzwerke direkt in unsere Shiny-Anwendung integrieren und sie mit einer stabilen Performance ausführen, wenn wir die Funktion visIgraphLayout verwenden. So benötigen wir keine externe Software wie Gephi mehr.
Habe ich dein Interesse geweckt, deine eigenen Netzwerke zu visualisieren? Dann kannst du gerne meinen Code verwenden oder mich kontaktieren und die Github-Seite des verwendeten Pakets hier besuchen.
Quellen
Knuth, D. E. (1993) „Les miserables: coappearance network of characters in the novel les miserables“, The Stanford GraphBase: A Platform for Combinatorial Computing, Addison-Wesley, Reading, MA
Seit der Veröffentlichung im Jahr 2014 hat sich Docker zu einem unverzichtbaren Werkzeug für die Bereitstellung von Anwendungen entwickelt. Bei STATWORX ist R eines unserer täglichen Werkzeuge, weshalb viele von uns begeistert sind von RStudio’s Rocker Projekt, das die Containerisierung von R-Code einfacher macht denn je.
Containerisierung ist in vielen verschiedenen Situationen nützlich. Die Technologie ist äußerst hilfreich beim Einsatz von R-Code in einer Cloud-Computing-Umgebung, in welcher der gecodete Arbeitsablauf in regelmäßigen Abständen ausgeführt werden soll. Docker ist für diese Aufgabe aus zwei Gründen perfekt geeignet: Container können automatisiert in gewünschten Intervallen gestartet werden und aufgrund ihrer statischen Natur ist immer klar, welches Verhalten und welchen Output du von einem Docker-Container zu erwarten hast. Wenn du also vor der Aufgabe stehst, ein Machine-Learning-Modell für regelmäßige Prognosen einzusetzen, dann lohnt es sich dies mit Docker tun. Dieser Blogbeitrag führt dich Schritt für Schritt durch den gesamten Prozess, wie du dein R-Skript in einem Docker-Container zum Laufen bringst. Der Einfachheit halber werden wir mit einem lokalen Datensatz arbeiten.
Zu Beginn möchte ich betonen, dass dieser Blogbeitrag kein allgemeines Docker-Tutorial ist. Wenn du dir nicht sicher bist was mit Images und Containern gemeint ist, dann empfehle ich dir, zunächst einen Blick auf das Docker Curriculum zu werfen. Solltest du daran interessiert sein, eine RStudio-Sitzung in einem Docker-Container laufen zu lassen, empfehle ich dir stattdessen dem OpenSciLabs Docker Tutorial einen Besuch abzustatten.
In diesem Blogbeitrag geht es konkret um die Containerisierung eines R-Skripts, damit es schließlich bei jedem Start des Containers automatisch ausgeführt wird, ohne dass der Benutzer eingreifen muss – damit entfällt die Notwendigkeit des RStudio-IDE. Ich werde nur kurz auf die im Dockerfile und in der Kommandozeile verwendete Syntax eingehen, so dass du dich am besten mit den Grundlagen von Docker bereits vor dem Weiterlesen vertraut machst.
Was wir brauchen:
Für diesen gesamten Workflow benötigen wir folgendes:
- ein R-Skript, das wir in ein Docker-Image einbauen
- ein Base-Image, auf dem wir unser eigenes Image aufbauen
- ein Dockerfile, mit der wir unser neues Image definieren
Du kannst alle Dateien und die verwendete Ordnerstruktur aus dem STATWORX GitHub Repository klonen.
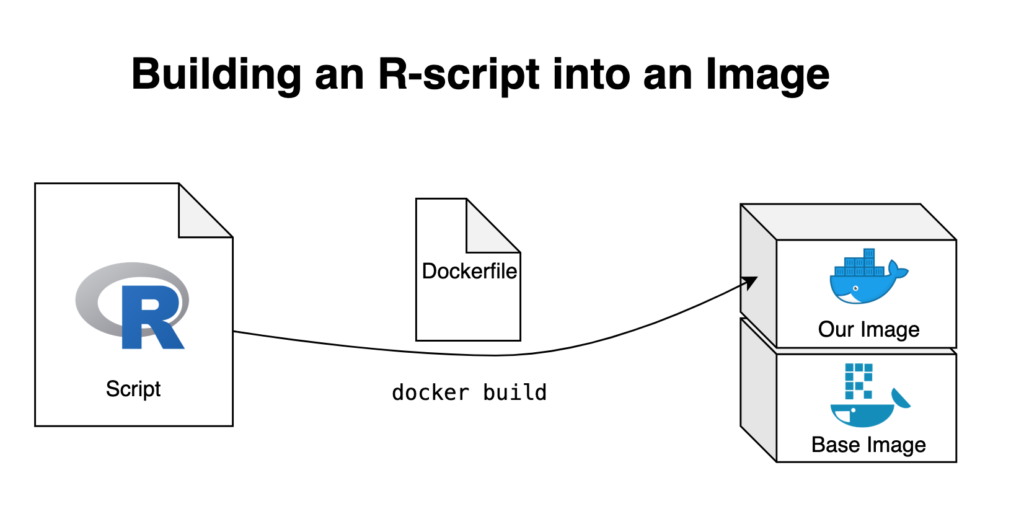
Das R-Skript
Wir arbeiten mit einem sehr einfachen R-Skript, das einen Datensatz importiert, als Dataframe manipuliert, ein Diagramm auf der Grundlage der manipulierten Daten erstellt und zum Schluss sowohl das Diagramm als auch die darauf basierenden Daten exportiert. Der für dieses Beispiel verwendete Datensatz ist US 500 Records, der von Brian Dunning zur Verfügung gestellt wird. Wenn du dem Beispiel folgen möchtest, empfehle ich dir, diesen Datensatz in den Ordner 01_data zu kopieren.
library(readr)
library(dplyr)
library(ggplot2)
library(forcats)
# import dataframe
df <- read_csv("01_data/us-500.csv")
# manipulate data
plot_data <- df %>%
group_by(state) %>%
count()
# save manipulated data to output folder
write_csv(plot_data, "03_output/plot_data.csv")
# create plot based on manipulated data
plot <- plot_data %>%
ggplot()+
geom_col(aes(fct_reorder(state, n),
n,
fill = n))+
coord_flip()+
labs(
title = "Number of people by state",
subtitle = "From US-500 dataset",
x = "State",
y = "Number of people"
)+
theme_bw()
# save plot to output folder
ggsave("03_output/myplot.png", width = 10, height = 8, dpi = 100)Damit wird ein einfaches Balkendiagramm auf der Grundlage des Datensatzes erstellt:
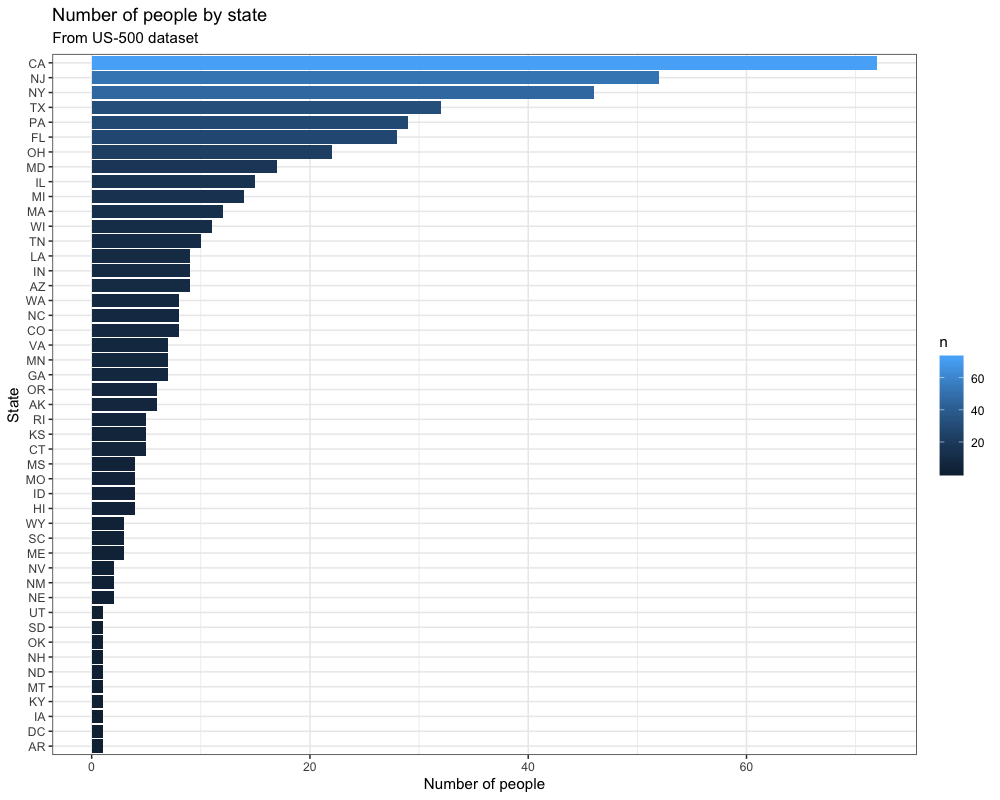
Wir verwenden dieses Skript, weil wir nicht nur R-Code innerhalb eines Docker-Containers ausführen, sondern diesen R-Code auf Daten von außerhalb unseres Containers anwenden und die Ergebnisse anschließend speichern wollen.
Das Base-Image
Die listet alle verfügbaren Rocker-Repositories auf. Da wir in unserem Skript Tidyverse-Pakete verwenden, ist das rocker/tidyverse-Image eine naheliegende Wahl. Das Problem mit diesem Repository ist, dass es auch RStudio selbst enthält, was wir für dieses spezifische Projekt nicht benötigen. Das bedeutet, dass wir stattdessen mit dem r-base Repository arbeiten und unser eigenes Tidyverse-fähiges Image erstellen. Wir können das rocker/r-base-Image von DockerHub nutzen, indem wir den folgenden Befehl im Terminal ausführen:
docker pull rocker/r-base
Dadurch wird das Base-R-Image aus dem Rocker DockerHub-Repository lokal gespeichert („gepullt“). Wir können einen auf diesem Image basierenden Container starten, indem wir im Terminal folgenden Bashcode ausführen:
docker run -it --rm rocker/r-base
-it interagieren können. Das Argument --rm sorgt dafür, dass der Container automatisch gelöscht wird, sobald wir ihn stoppen. Es steht dir frei, mit deiner containerisierten R-Sitzung zu experimentieren (die du mit der Funktion q() in der R-Konsole wieder beenden kannst). Du kannst zum Beispiel damit beginnen, die Pakete, die du für deinen Arbeitsablauf benötigst, mit install.packages()
Das Dockerfile
Mit einem Dockerfile teilen wir Docker mit, wie unser gewünschtes Image erstellt werden soll. Ein Dockerfile ist eine Textdatei, die „Dockerfile.txt“ heißen muss und standardmäßig im Stammverzeichnis des „Build-Kontextes“ liegt (in unserem Fall wäre das der Ordner „R-Script in Docker“).
Zunächst legen wir ein bestehendes Docker-Image fest, basierend auf dem wir unser neues Image erstellen möchten. Anschließend verfassen wir eine Liste von Anweisungen, die unser Image so definieren, dass die Ausführung von Containern reibungslos und effizient verläuft. In unserem Fall möchte ich unser neues Image auf dem zuvor besprochenen rocker/r-base-Image aufbauen. Ebenfalls möchte ich auch die lokale Ordnerstruktur replizieren, also erstelle ich die gewünschten Verzeichnisse direkt mit dem Dockerfile. Danach werden alle Dateien, auf die das Image Zugriff haben soll, in diese Verzeichnisse kopiert – so wird das R-Skript in das Docker-Image eingebaut. Auf diese Weise können wir auch die manuelle Installation von Paketen nach dem Starten eines Containers umgehen, da wir ein zweites R-Skript vorbereiten können, das sich um die Paketinstallation kümmert. Es reicht nicht aus, das R-Skript einfach zu kopieren, wir müssen Docker auch anweisen, es beim Erstellen des Images automatisch auszuführen. Und das ist unser erstes Dockerfile:
# Base image https://hub.docker.com/u/rocker/
FROM rocker/r-base:latest
## create directories
RUN mkdir -p /01_data
RUN mkdir -p /02_code
RUN mkdir -p /03_output
## copy files
COPY /02_code/install_packages.R /02_code/install_packages.R
COPY /02_code/myScript.R /02_code/myScript.R
## install R-packages
RUN Rscript /02_code/install_packages.Rinstall_packages.R
install.packages("readr")
install.packages("dplyr")
install.packages("ggplot2")
install.packages("forcats")Erstellen und Ausführen des Images
Nun haben wir alle benötigten Komponenten für unser neues Docker-Image vorbereitet. Verwende das Terminal, um zu dem Ordner zu navigieren, in dem sich dein Dockerfile befindet und erstelle das Image mit:
docker build -t myname/myimage .Der Prozess wird aufgrund der Paketinstallation einen Moment dauern. Sobald er abgeschlossen ist, kannst du das neue Image testen, indem du einen Container startest mit:
docker run -it --rm -v ~/"R-Script in Docker"/01_data:/01_data -v ~/"R-Script in Docker"/03_output:/03_output myname/myimageDie Verwendung der -v-Argumente teilt Docker mit, welche lokalen Ordner den erstellten Ordnern innerhalb des Containers zugeordnet werden sollen. Dies ist wichtig, da wir so sowohl Zugriff auf unseren Datensatz von innerhalb des Containers erhalten, als auch den erstellen Output des Workflows lokal abspeichern können. Dadurch muss der Datensatz nicht in das Image eingebaut werden und wenn das Image gestoppt wird, gehen keine Outputs verloren.
Dieser Container kann nun mit dem Datensatz im Ordner 01_data interagieren und hat eine Kopie unseres Workflow-Skripts in seinem eigenen Ordner 02_code. Wenn Sie R anweisen, source("02_code/myScript.R") auszuführen, wird das Skript ausgeführt und der Output im Ordner 03_output gespeichert, von wo aus er auch in den lokalen Ordner 03_output kopiert wird.
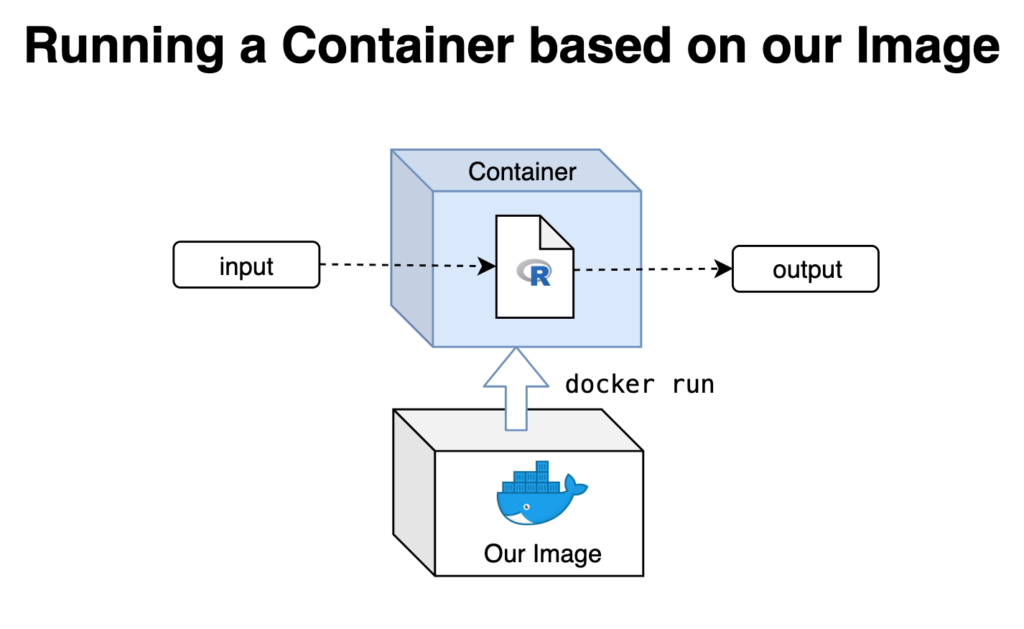
Verbessern was wir haben
Nachdem wir nun getestet und bestätigt haben, dass unser R-Skript im Container wie erwartet läuft, fehlen nur noch ein paar wenige Dinge.
- Wir wollen das Skript nicht manuell aus dem Container heraus aufrufen, sondern es automatisch ausführen lassen, sobald der Container gestartet wird.
Dies können wir ganz einfach erreichen, indem wir den folgenden Befehl an das Ende unserer Dockerdatei anhängen:
## run the script
CMD Rscript /02_code/myScript.RDas verweist auf den Speicherort unseres Skripts in der Ordnerstruktur des Containers, markiert es als R-Code und weist Docker dann an, beim Starten des Containers das Skript gleich auszuführen. Änderungen am Dockerfile bedeuten natürlich, dass wir unser Image neu erstellen müssen, und das wiederum bedeutet, dass der langsame Prozess der Paketinstallationen wiederholt werden muss. Das ist mühsam, vor allem wenn die Wahrscheinlichkeit besteht, dass es im Laufe der Zeit weitere Überarbeitungen der einzelnen Komponenten unseres Images geben wird. Deshalb schlage ich folgendes vor:
- Erstelle ein Docker-Zwischen-Image, auf dem alle wichtigen Pakete und Abhängigkeiten installiert sind. Auf diesem Zwischen-Image als Basis bauen wir dann unser gewünschtes, finales Image auf.
Auf diese Weise wird die Paketinstallation vom eigentlichen Image entkoppelt, so dass unser finales Image innerhalb von Sekunden neu gebaut werden kann. Dies ermöglicht es uns, frei mit dem Code zu experimentieren, ohne dass wir uns immer wieder mit der Installation von Paketen durch Docker beschäftigen müssen.
Erstellen eines Zwischen-Images
Das Dockerfile für unser Zwischen-Image sieht unserem vorherigen Beispiel sehr ähnlich. Da ich mich entschieden habe, das install_packages()-Skript zu modifizieren, um das gesamte tidyverse für die zukünftige Verwendung einzuschließen, müssen auch einige Debian-Pakete installiert werden, die das tidyverse benötigt. Nicht alle davon sind absolut notwendig, aber alle sind auf die eine oder andere Weise nützlich.
# Base image https://hub.docker.com/u/rocker/
FROM rocker/r-base:latest
# system libraries of general use
## install debian packages
RUN apt-get update -qq && apt-get -y --no-install-recommends install \
libxml2-dev \
libcairo2-dev \
libsqlite3-dev \
libmariadbd-dev \
libpq-dev \
libssh2-1-dev \
unixodbc-dev \
libcurl4-openssl-dev \
libssl-dev
## update system libraries
RUN apt-get update && \
apt-get upgrade -y && \
apt-get clean
## create directories
RUN mkdir -p /02_code
## copy files
COPY /02_code/install_packages.R /02_code/install_packages.R
## install R-packages
RUN Rscript /02_code/install_packages.RIch baue das Image, indem ich im Terminal zu dem Ordner, in dem sich mein Dockerfile befindet, navigiere und den Befehl docker build
docker build -t oliverstatworx/base-r-tidyverse .Ich habe dieses Image auch auf gepusht. So kannst du, wenn du jemals ein Base-R-Image mit vorinstalliertem tidyverse benötigst, es einfach auf meinem Image aufbauen, ohne dieses selbst erstellen zu müssen.
Nun, da das Zwischen-Image erstellt wurde, können wir unser ursprüngliches Dockerfile so ändern, dass es anstelle von rocker/r-base darauf aufbaut. Da sich unser Zwischen-Image bereits um die Paketinstallation kümmert kann dieser Abschnitt entfernt werden. Wir fügen auch die letzte Zeile hinzu, die unser Skript automatisch ausführt, sobald der Container gestartet wird. Unser endgültiges Dockerfile sollte in etwa so aussehen:
# Base image https://hub.docker.com/u/oliverstatworx/
FROM oliverstatworx/base-r-tidyverse:latest
## create directories
RUN mkdir -p /01_data
RUN mkdir -p /02_code
RUN mkdir -p /03_output
## copy files
COPY /02_code/myScript.R /02_code/myScript.R
## run the script
CMD Rscript /02_code/myScript.RDer letzte Schliff
Da wir unser Image auf einem Zwischen-Image mit all unseren benötigten Paketen aufgebaut haben, können wir nun beliebig Teile des Dockerfiles ohne großen Zeitaufwand verändern. Beispielsweise kann es sinnvoll sein, das R-Skript so zu gestalten, dass Warnungen und Meldungen, die nicht mehr von Interesse sind (da das Image bereits getestet wurde und alles wie erwartet funktioniert) unterdrückt werden. Des weiteren können Meldungen hinzufügt werden, die dem Benutzenden mitteilen, welcher Teil des Skripts gerade von dem laufenden Container ausgeführt wird.
suppressPackageStartupMessages(library(readr))
suppressPackageStartupMessages(library(dplyr))
suppressPackageStartupMessages(library(ggplot2))
suppressPackageStartupMessages(library(forcats))
options(scipen = 999,
readr.num_columns = 0)
print("Starting Workflow")
# import dataframe
print("Importing Dataframe")
df <- read_csv("01_data/us-500.csv")
# manipulate data
print("Manipulating Data")
plot_data <- df %>%
group_by(state) %>%
count()
# save manipulated data to output folder
print("Writing manipulated Data to .csv")
write_csv(plot_data, "03_output/plot_data.csv")
# create plot based on manipulated data
print("Creating Plot")
plot <- plot_data %>%
ggplot()+
geom_col(aes(fct_reorder(state, n),
n,
fill = n))+
coord_flip()+
labs(
title = "Number of people by state",
subtitle = "From US-500 dataset",
x = "State",
y = "Number of people"
)+
theme_bw()
# save plot to output folder
print("Saving Plot")
ggsave("03_output/myplot.png", width = 10, height = 8, dpi = 100)
print("Worflow Finished")docker build -t myname/myimage . Erneut starten wir einen Container auf Basis unseres Images und weisen die Ordner 01_data und 03_output
docker run -it --rm -v ~/"R-Script in Docker"/01_data:/01_data -v ~/"R-Script in Docker"/03_output:/03_output myname/myimageHerzlichen Glückwunsch, du hast jetzt ein sauberes Docker-Image erstellt! Dieses führt beim Containerstart nicht nur automatisch dein R-Skript aus, sondern teilt dirauch über Konsolenmeldungen genau mit, welchen Teil des Codes gerade ausgeführt wird. Viel Spaß beim Dockern!